DMR 136 chr1:16275301-16275600 (300bp)
| DMR Stats | |
|---|---|
| Position | chr1:16275301-16275600 (View on UCSC) |
| Width | 300bp |
| Genomic Context | intron |
| Nearest Genes | ZBTB17 (0bp away) |
| ANODEV p-value | 2.83214622935e-05 |
| Frequencies | Benign: 0.0 (0.0%), Low: 0.0 (0.0%), High: 0.0 (0.0 %) |
| Frequent? |
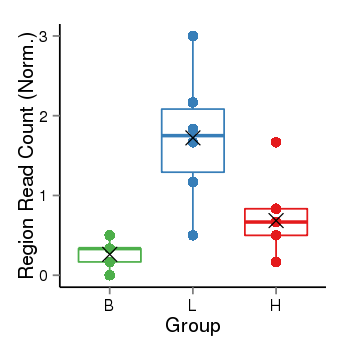
Region Count Boxplot
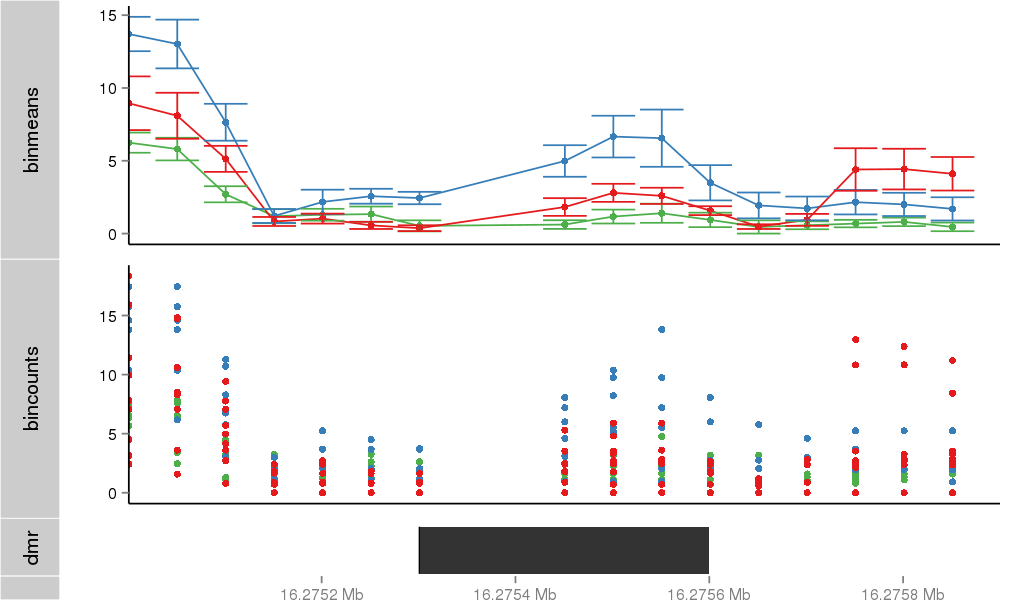
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 136 | ZBTB17 | 288 | uc001axl.4 | chr1 | 16268364 | 16302627 | - | 300 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc009vom.1 | chr1 | 16271432 | 16302627 | - | 300 | 100.0 | 0.09 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc009von.1 | chr1 | 16274238 | 16302627 | - | 300 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (100), E2 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obq.2 | chr1 | 16268364 | 16302627 | - | 300 | 100.0 | 0.05 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obr.2 | chr1 | 16268364 | 16302627 | - | 300 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obs.2 | chr1 | 16268364 | 16302627 | - | 300 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obt.1 | chr1 | 16270322 | 16302627 | - | 300 | 100.0 | 0.07 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obu.2 | chr1 | 16270365 | 16302627 | - | 300 | 100.0 | 0.05 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0) | 0.0 |
| 136 | ZBTB17 | 288 | uc010obv.1 | chr1 | 16272257 | 16302627 | - | 300 | 100.0 | 0.07 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0) | 0.0 |
- 9 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 136 | chr1 | 16275106 | 16275415 | wgEncodeRegDnaseClusteredV2 | 19 | score:502, srow:12468 |
| 136 | chr1 | 16275426 | 16275915 | wgEncodeRegDnaseClusteredV2 | 85 | score:1000, srow:12469 |
| 136 | chr1 | 16275033 | 16275552 | wgEncodeRegTfbsClusteredV3 | ATF2 | score:369, expCount:1, expNums:79, expScores:369, srow:41072 |
| 136 | chr1 | 16275163 | 16275452 | wgEncodeRegTfbsClusteredV3 | MEF2A | score:183, expCount:1, expNums:93, expScores:183, srow:41073 |
| 136 | chr1 | 16275199 | 16275434 | wgEncodeRegTfbsClusteredV3 | BATF | score:168, expCount:1, expNums:81, expScores:168, srow:41074 |
| 136 | chr1 | 16275210 | 16275362 | wgEncodeRegTfbsClusteredV3 | SRF | score:1000, expCount:1, expNums:113, expScores:1000, srow:41075 |
| 136 | chr1 | 16275265 | 16275634 | wgEncodeRegTfbsClusteredV3 | RELA | score:192, expCount:1, expNums:325, expScores:192, srow:41076 |
| 136 | chr1 | 16275393 | 16275861 | wgEncodeRegTfbsClusteredV3 | CTCF | score:374, expCount:11, expNums:16,18,35,37,44,643,646,657,659,671,672, expScores:151,221,100,146,197,374,157,126,186,143,117, srow:41077 |
| 136 | chr1 | 16275563 | 16275918 | wgEncodeRegTfbsClusteredV3 | MAX | score:170, expCount:1, expNums:511, expScores:170, srow:41078 |
| 136 | chr1 | 16275582 | 16275945 | wgEncodeRegTfbsClusteredV3 | TEAD4 | score:260, expCount:1, expNums:240, expScores:260, srow:41079 |
| 136 | chr1 | 16275594 | 16275902 | wgEncodeRegTfbsClusteredV3 | STAT3 | score:632, expCount:5, expNums:565,566,567,568,569, expScores:123,316,390,374,632, srow:41080 |
| 136 | chr1 | 16273583 | 16275582 | cpgShelf | — | values(x.gr)[overs$srow, ]:623 |
- 12 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 136 | cellmeth_recounts | PrEC: 0, LNCaP: 0, DU145: 0 |  |
| 136 | cellexp | id=ENSG00000116809, name=ZBTB17, PrEC=1483, str=1406, LNCaP=989, str=679, DU145=1203, str=954 | 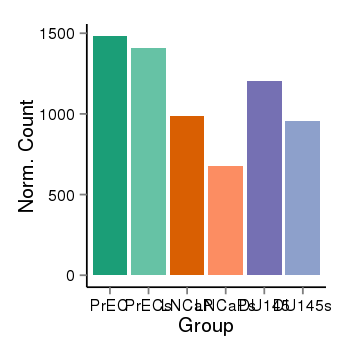 |
| 136 | tcgaexp | gene=ZBTB17, entrez=7709, pos=chr1:16268364-16302627(-), B=1257, L=1520, M=1420, H=1337 | 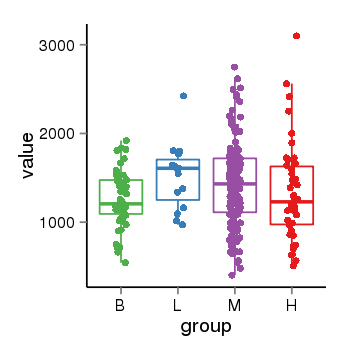 |
| 136 | tcgaexp | gene=SRSF10, entrez=10772, pos=chr1:36273-24306953(-), B=3487, L=3388, M=3432, H=4019 | 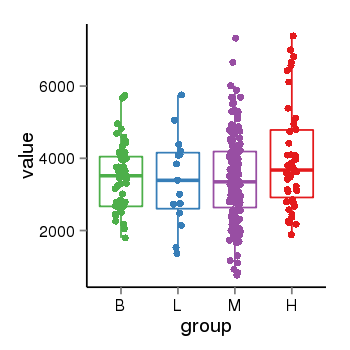 |
| 136 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |