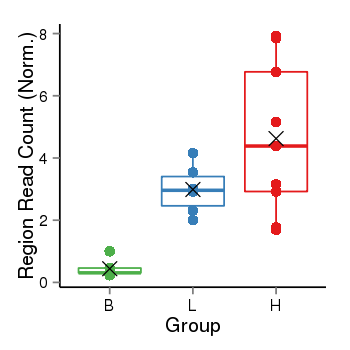
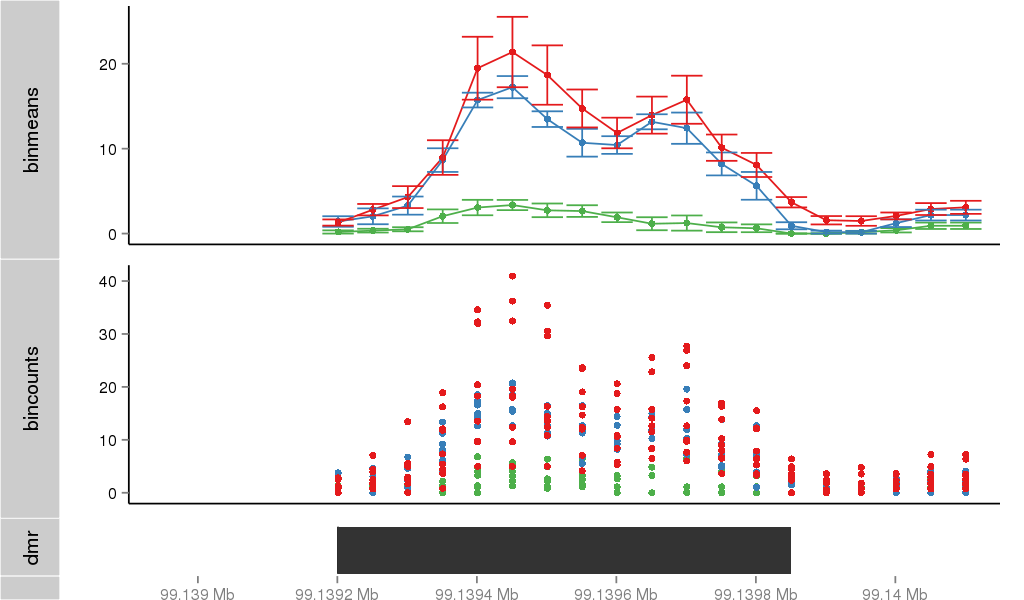
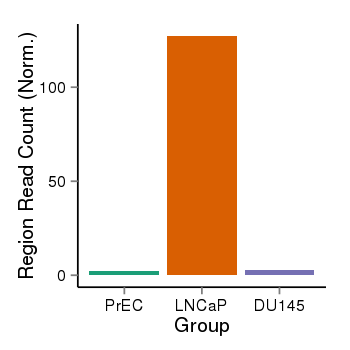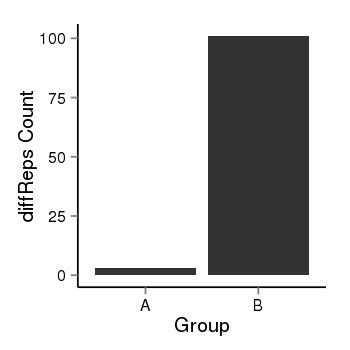| 32267 | ANKS1B | 6897 | uc001tgd.2 | chr12 | 99128569 | 99548577 | - | 650 | 100.0 | 0.24 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (100), E11 (0) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tge.2 | chr12 | 99128569 | 100378432 | - | 650 | 100.0 | 0.35 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (100), E26 (0) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgf.2 | chr12 | 99128569 | 100378432 | - | 650 | 100.0 | 0.46 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (100), E18 (0) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgg.4 | chr12 | 99137752 | 99548645 | - | 650 | 100.0 | 0.35 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (34.92), E12 (65.23) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgh.4 | chr12 | 99137752 | 99288674 | - | 650 | 100.0 | 0.63 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (34.46), E10 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgi.3 | chr12 | 99137752 | 99548577 | - | 650 | 100.0 | 1.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (34.46), E13 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgj.3 | chr12 | 99137752 | 99548577 | - | 650 | 100.0 | 0.83 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (34.46), E11 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc001tgk.3 | chr12 | 99137752 | 99640642 | - | 650 | 100.0 | 0.51 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (34.46), E14 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc009ztp.3 | chr12 | 99137752 | 99288674 | - | 650 | 100.0 | 0.63 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (34.92), E11 (65.23) | 0.0 |
| 32267 | ANKS1B | 6897 | uc009ztr.3 | chr12 | 99137752 | 99548577 | - | 650 | 100.0 | 0.38 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (34.46), E12 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc009zts.2 | chr12 | 99137752 | 99548577 | - | 650 | 100.0 | 0.38 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (32), E12 (68.15) | 0.0 |
| 32267 | ANKS1B | 6897 | uc010svd.2 | chr12 | 99128569 | 99288674 | - | 650 | 100.0 | 0.28 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (100), E10 (0) | 0.0 |
| 32267 | ANKS1B | 6897 | uc010sve.2 | chr12 | 99137752 | 99288674 | - | 650 | 100.0 | 0.14 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (34.46), E11 (65.69) | 0.0 |
| 32267 | ANKS1B | 6897 | uc010svf.2 | chr12 | 99137752 | 99288674 | - | 650 | 100.0 | 0.11 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (34.92), E11 (65.23) | 0.0 |
| 32267 | ANKS1B | 6897 | uc010svg.2 | chr12 | 99137752 | 99548868 | - | 650 | 100.0 | 0.14 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (34.92), E12 (65.23) | 0.0 |