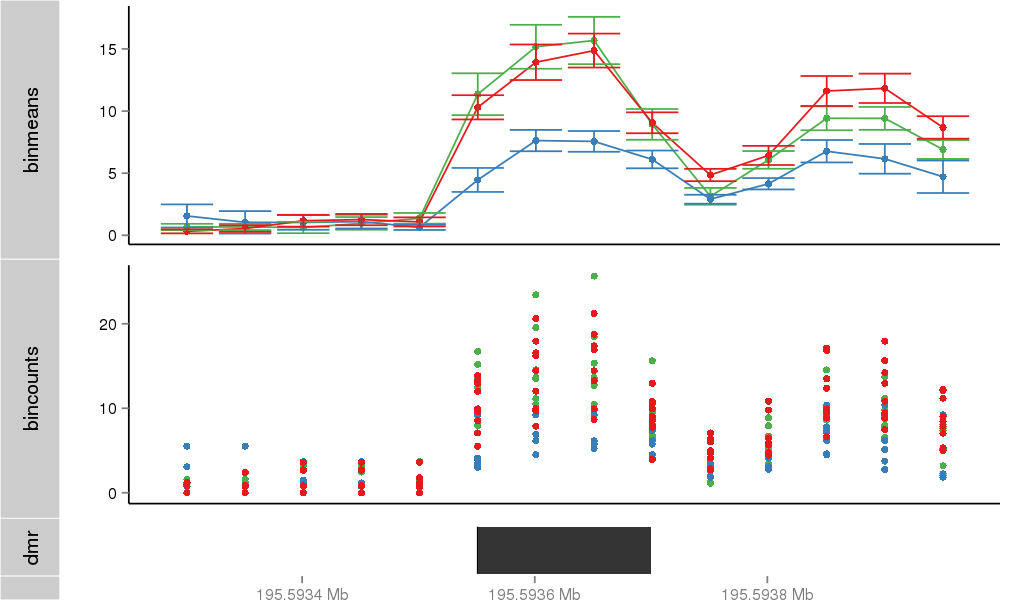
DMR 13266 chr3:195593551-195593700 (150bp)
| DMR Stats | |
|---|---|
| Position | chr3:195593551-195593700 (View on UCSC) |
| Width | 150bp |
| Genomic Context | 3' end |
| Nearest Genes | TNK2 (0bp away) |
| ANODEV p-value | 0.000656820430809 |
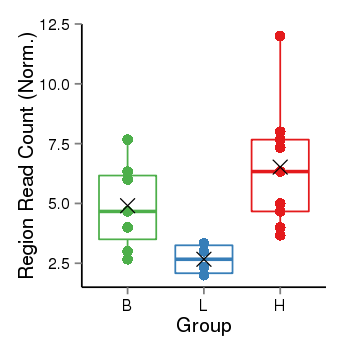
| Frequencies | Benign: 5.0 (71.43%), Low: 2.0 (33.33%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 13266 | TNK2 | 21199 | uc003fvq.1 | chr3 | 195590236 | 195596561 | - | 150 | 100.0 | 0.11 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (100), E4 (0) | 0.0 |
| 13266 | TNK2 | 21199 | uc003fvr.1 | chr3 | 195590236 | 195599341 | - | 150 | 100.0 | 0.16 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (100), E7 (0) | 0.0 |
| 13266 | TNK2 | 21199 | uc003fvs.1 | chr3 | 195590236 | 195619646 | - | 150 | 100.0 | 0.16 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (100), E14 (0) | 0.0 |
| 13266 | TNK2 | 21199 | uc003fvt.1 | chr3 | 195590236 | 195622432 | - | 150 | 100.0 | 0.82 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (100), E15 (0) | 0.0 |
| 13266 | TNK2 | 21199 | uc003fvu.1 | chr3 | 195590236 | 195635880 | - | 150 | 100.0 | 0.24 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (100), E15 (0) | 0.0 |
- 5 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 13266 | chr3 | 195593606 | 195593895 | wgEncodeRegDnaseClusteredV2 | 7 | score:418, srow:862604 |
| 13266 | chr3 | 195592837 | 195594836 | cpgShore | — | values(x.gr)[overs$srow, ]:34878 |
- 2 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 13266 | cellmeth_recounts | PrEC: 30, LNCaP: 9, DU145: 10 | 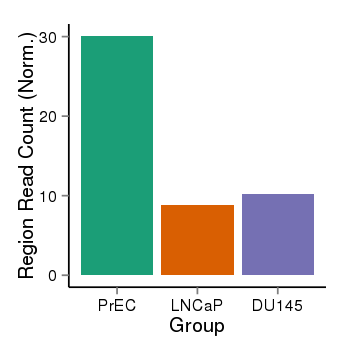 |
| 13266 | cellmeth_diffreps_PrECvsLNCaP | A=45, B=9, Length=380, Event=Down, log2FC=-2.32, padj=1.66451854270562e-05 | 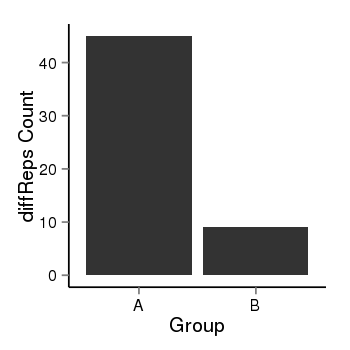 |
| 13266 | cellmeth_diffreps_PrECvsDU145 | A=78, B=11, Length=620, Event=Down, log2FC=-2.83, padj=6.85813601030989e-12 | 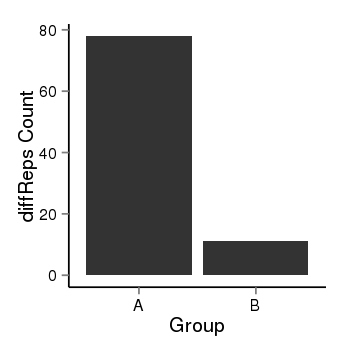 |
| 13266 | cellexp | id=ENSG00000061938, name=TNK2, PrEC=4753, str=2687, LNCaP=6220, str=2874, DU145=4002, str=2747 | 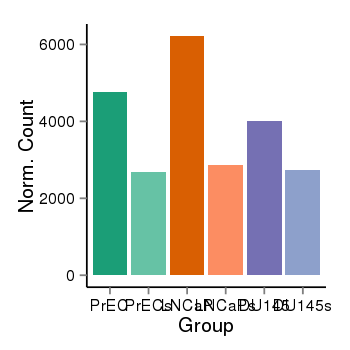 |
| 13266 | tcgaexp | gene=TNK2, entrez=10188, pos=chr3:195590236-195638816(-), B=2766, L=4388, M=4178, H=4627 | 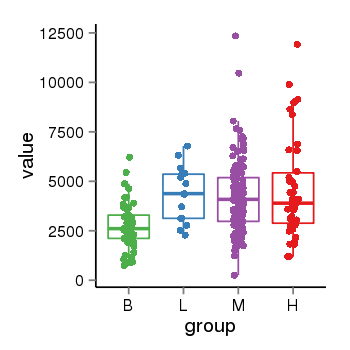 |