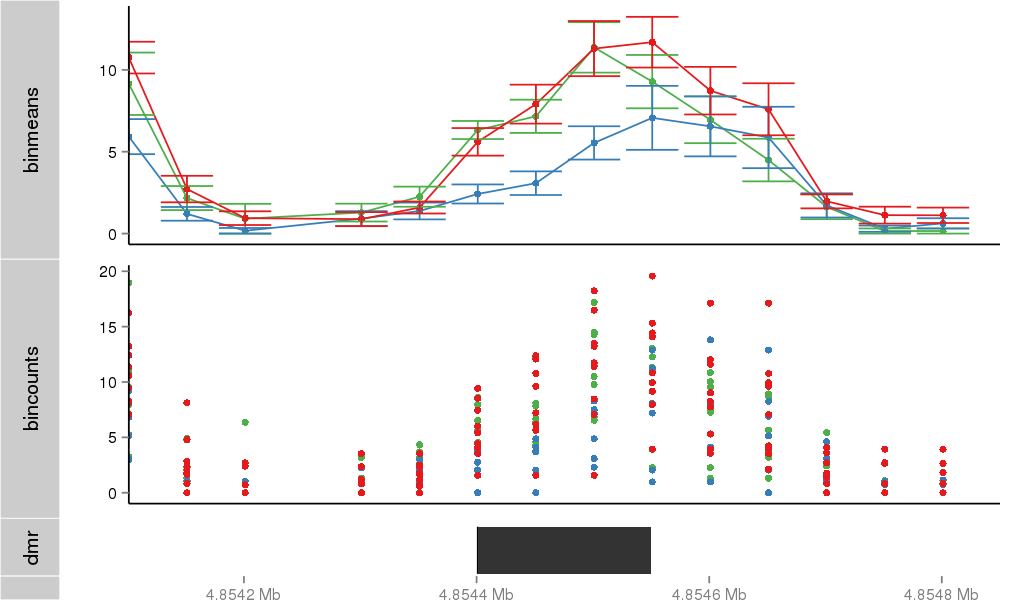
DMR 21078 chr16:4854401-4854550 (150bp)
| DMR Stats | |
|---|---|
| Position | chr16:4854401-4854550 (View on UCSC) |
| Width | 150bp |
| Genomic Context | 3' end |
| Nearest Genes | GLYR1 (0bp away) |
| ANODEV p-value | 0.0267383492297 |
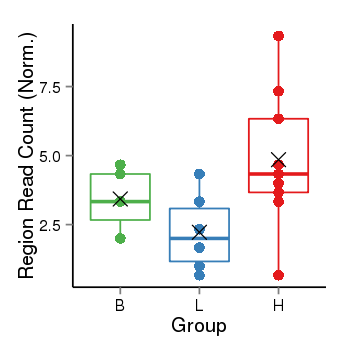
| Frequencies | Benign: 5.0 (71.43%), Low: 2.0 (33.33%), High: 8.0 (88.89 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 21078 | GLYR1 | 10909 | uc002cxx.4 | chr16 | 4853204 | 4897303 | - | 150 | 100.0 | 0.69 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (100) | 0.0 |
| 21078 | GLYR1 | 10909 | uc002cxy.3 | chr16 | 4853204 | 4897303 | - | 150 | 100.0 | 0.77 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (18.67), I17 (81.33), E18 (0) | 0.0 |
| 21078 | GLYR1 | 10909 | uc002cxz.1 | chr16 | 4854523 | 4896211 | - | 28 | 18.67 | 0.96 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (18.67) | 100.0 |
| 21078 | GLYR1 | 10909 | uc002cya.2 | chr16 | 4854523 | 4897303 | - | 28 | 18.67 | 0.96 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (18.67) | 100.0 |
| 21078 | GLYR1 | 10909 | uc010uxv.1 | chr16 | 4854523 | 4897303 | - | 28 | 18.67 | 0.58 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (18.67) | 100.0 |
- 5 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 21078 | chr16 | 4853966 | 4854635 | wgEncodeRegDnaseClusteredV2 | 18 | score:299, srow:431540 |
| 21078 | chr16 | 4854345 | 4855068 | wgEncodeRegTfbsClusteredV3 | RBBP5 | score:260, expCount:1, expNums:33, expScores:260, srow:1461452 |
| 21078 | chr16 | 4854414 | 4854426 | tfbsConsSites | V$OCT_C | score:833, zScore:2.2, srow:1882072 |
| 21078 | chr16 | 4854402 | 4854428 | phastConsElements100way | lod=95 | score:444, srow:3240580 |
| 21078 | chr16 | 4853177 | 4855176 | cpgShore | — | values(x.gr)[overs$srow, ]:16110 |
- 5 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 21078 | cellmeth_recounts | PrEC: 10, LNCaP: 49, DU145: 5 | 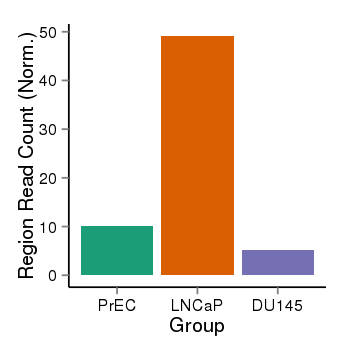 |
| 21078 | cellmeth_diffreps_PrECvsLNCaP | A=7, B=35, Length=200, Event=Up, log2FC=2.32, padj=0.000277572010749213 | 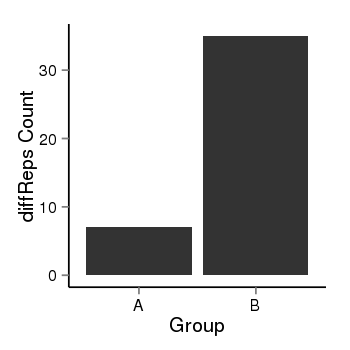 |
| 21078 | cellmeth_diffreps_PrECvsDU145 | A=11, B=0, Length=200, Event=Down, log2FC=-Inf, padj=0.00313959514768542 | 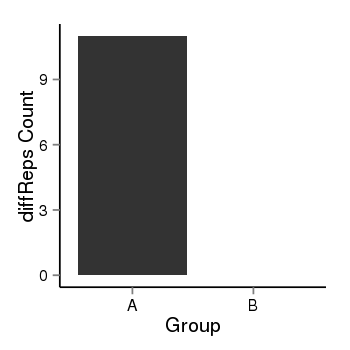 |
| 21078 | cellmeth_diffreps_LNCaPvsDU145 | A=121.87, B=10.29, Length=940, Event=Down, log2FC=-3.57, padj=0 | 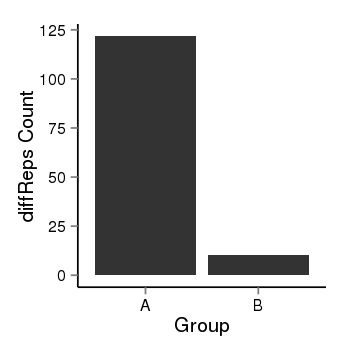 |
| 21078 | cellexp | id=ENSG00000140632, name=GLYR1, PrEC=3875, str=2493, LNCaP=5261, str=5363, DU145=2452, str=2164 | 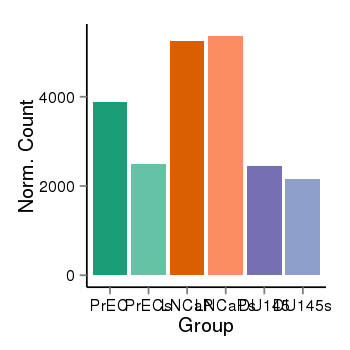 |
| 21078 | cellexp | id=ENSG00000256599, name=AC020663.1, PrEC=0, str=0, LNCaP=0, str=0, DU145=0, str=0 |  |
| 21078 | tcgaexp | gene=GLYR1, entrez=84656, pos=chr16:4853204-4897303(-), B=8134, L=8110, M=7658, H=7873 | 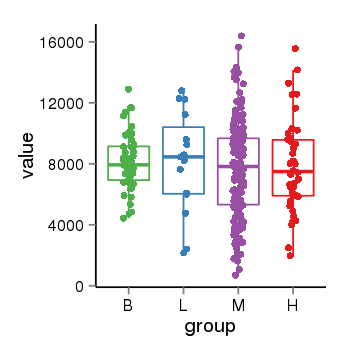 |