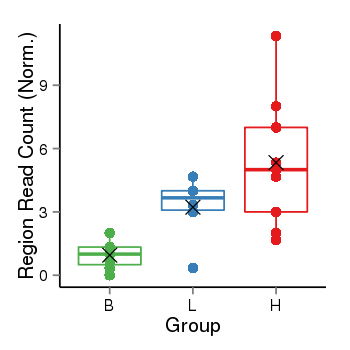
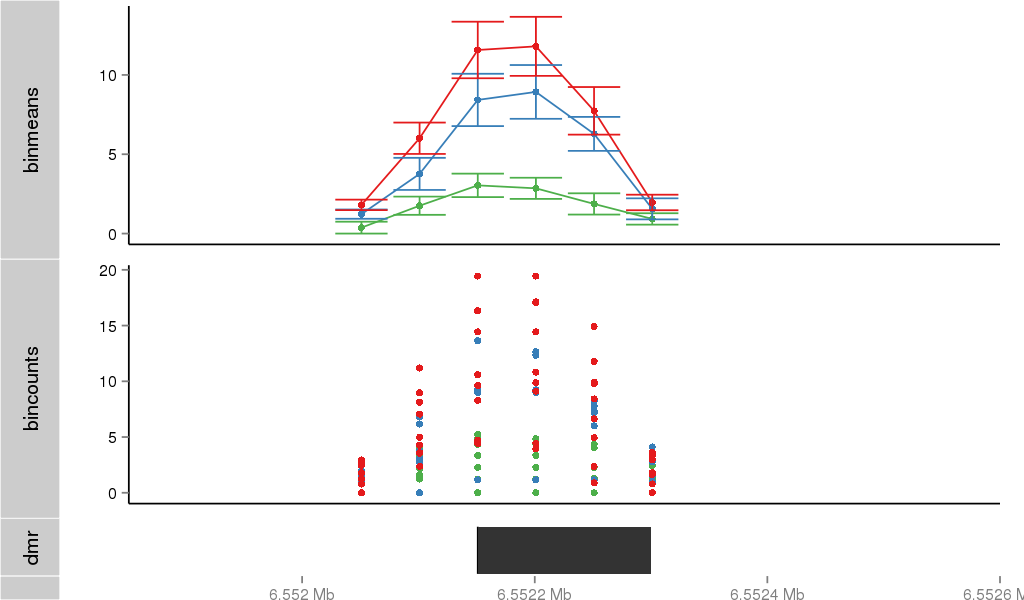
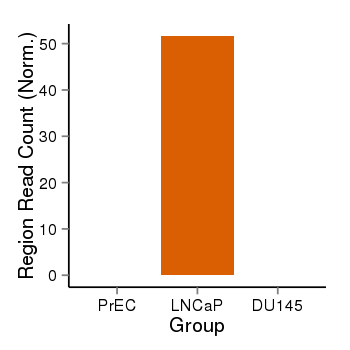
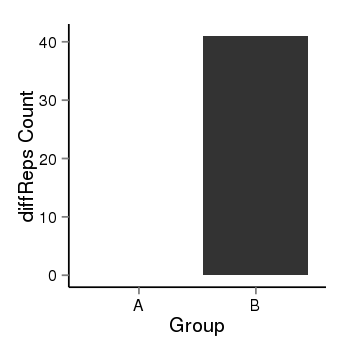| 24948 | PLEKHG5 | 154 | uc001ann.1 | chr1 | 6526152 | 6556756 | - | 150 | 100.0 | 0.51 | 0 | 0.0 | E1 (0), I1 (100), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0) | 0.0 |
| 24948 | PLEKHG5 | 154 | uc001ano.1 | chr1 | 6526152 | 6557484 | - | 150 | 100.0 | 0.3 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |
| 24948 | PLEKHG5 | 154 | uc001anp.2 | chr1 | 6526152 | 6580121 | - | 150 | 100.0 | 0.3 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |
| 24948 | PLEKHG5 | 154 | uc001anq.2 | chr1 | 6526152 | 6580121 | - | 150 | 100.0 | 0.08 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |
| 24948 | PLEKHG5 | 154 | uc031plb.1 | chr1 | 6526152 | 6557156 | - | 150 | 100.0 | 0.08 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |