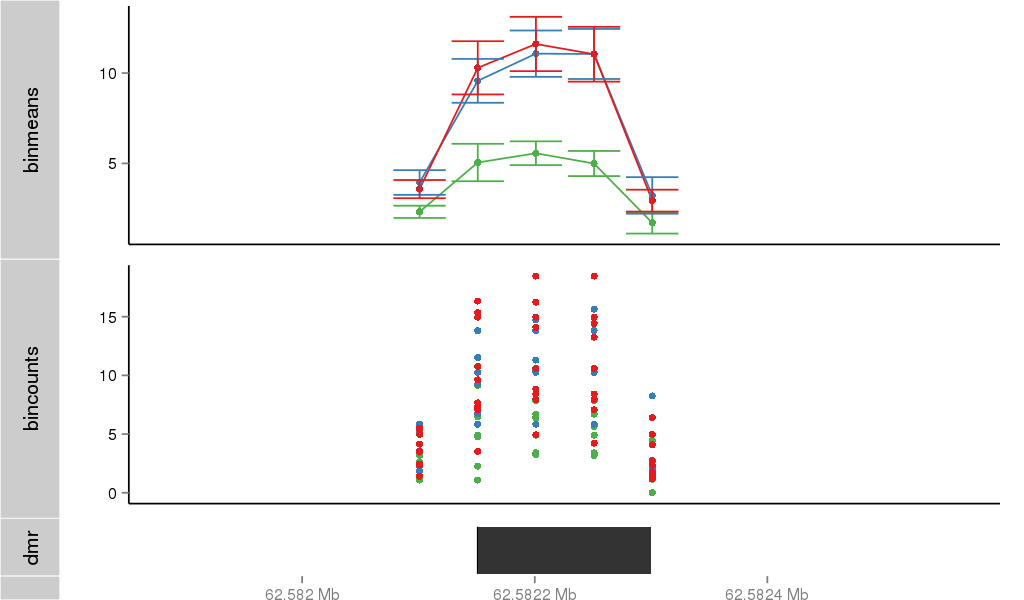
DMR 25399 chr1:62582151-62582300 (150bp)
| DMR Stats | |
|---|---|
| Position | chr1:62582151-62582300 (View on UCSC) |
| Width | 150bp |
| Genomic Context | 3' end |
| Nearest Genes | INADL (0bp away) |
| ANODEV p-value | 0.00360048517149 |
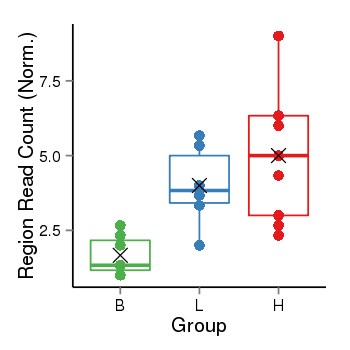
| Frequencies | Benign: 0.0 (0.0%), Low: 5.0 (83.33%), High: 6.0 (66.67 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 25399 | INADL | 964 | uc001dab.3 | chr1 | 62208149 | 62629591 | + | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0), I27 (0), E28 (0), I28 (0), E29 (0), I29 (0), E30 (0), I30 (0), E31 (0), I31 (0), E32 (0), I32 (0), E33 (0), I33 (0), E34 (0), I34 (0), E35 (0), I35 (35.33), E36 (64.67), I36 (0), E37 (0), I37 (0), E38 (0), I38 (0), E39 (0), I39 (0), E40 (0), I40 (0), E41 (0), I41 (0), E42 (0), I42 (0), E43 (0) | 0.0 |
| 25399 | INADL | 964 | uc001dac.3 | chr1 | 62271120 | 62629176 | + | 150 | 100.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (35.33), E24 (64.67), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0), I27 (0), E28 (0), I28 (0), E29 (0), I29 (0), E30 (0), I30 (0), E31 (0), I31 (0), E32 (0), I32 (0), E33 (0) | 0.0 |
| 25399 | INADL | 964 | uc009wag.3 | chr1 | 62417926 | 62644347 | + | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (35.33), E10 (64.67), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 25399 | INADL | 964 | uc010oot.2 | chr1 | 62417926 | 62583006 | + | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (35.33), E11 (64.67), I11 (0), E12 (0) | 100.0 |
- 4 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 25399 | chr1 | 62582099 | 62582508 | wgEncodeRegTfbsClusteredV3 | GATA2 | score:217, expCount:1, expNums:586, expScores:217, srow:159407 |
| 25399 | chr1 | 62582214 | 62582229 | tfbsConsSites | V$FREAC2_01 | score:833, zScore:2.09, srow:157751 |
| 25399 | chr1 | 62582246 | 62582264 | tfbsConsSites | V$SEF1_C | score:748, zScore:2.22, srow:157752 |
| 25399 | chr1 | 62582246 | 62582269 | tfbsConsSites | V$BRACH_01 | score:683, zScore:1.76, srow:157753 |
| 25399 | chr1 | 62582291 | 62582307 | tfbsConsSites | V$YY1_01 | score:864, zScore:1.76, srow:157754 |
| 25399 | chr1 | 62582244 | 62582244 | cosmic | COSM240229 | srow:36664 |
| 25399 | chr1 | 62582249 | 62582249 | cosmic | COSM1651210 | srow:36665 |
| 25399 | chr1 | 62582249 | 62582249 | cosmic | COSM911293 | srow:36666 |
| 25399 | chr1 | 62582250 | 62582250 | cosmic | COSM245032 | srow:36667 |
| 25399 | chr1 | 62582282 | 62582282 | cosmic | COSM1343759 | srow:36668 |
| 25399 | chr1 | 62582288 | 62582288 | cosmic | COSM261421 | srow:36669 |
| 25399 | chr1 | 62582171 | 62582172 | phastConsElements100way | lod=13 | score:247, srow:272642 |
| 25399 | chr1 | 62582185 | 62582211 | phastConsElements100way | lod=156 | score:493, srow:272643 |
| 25399 | chr1 | 62582214 | 62582248 | phastConsElements100way | lod=395 | score:585, srow:272644 |
| 25399 | chr1 | 62582250 | 62582257 | phastConsElements100way | lod=74 | score:420, srow:272645 |
| 25399 | chr1 | 62582259 | 62582263 | phastConsElements100way | lod=81 | score:429, srow:272646 |
| 25399 | chr1 | 62582265 | 62582314 | phastConsElements100way | lod=525 | score:613, srow:272647 |
- 17 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 25399 | cellmeth_recounts | PrEC: 1, LNCaP: 0, DU145: 0 | 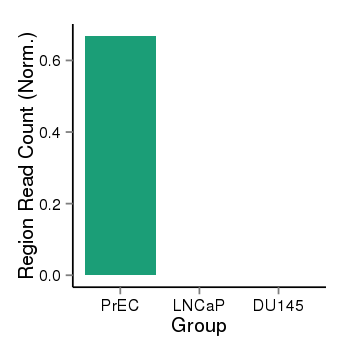 |
| 25399 | cellexp | id=ENSG00000132849, name=INADL, PrEC=4804, str=7225, LNCaP=3288, str=4886, DU145=3326, str=3971 | 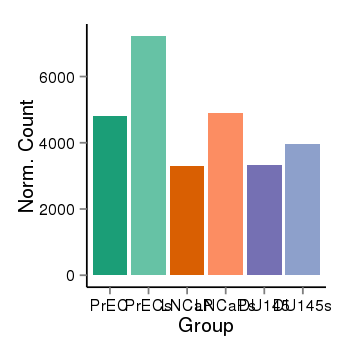 |
| 25399 | tcgaexp | gene=INADL, entrez=10207, pos=chr1:62208149-62644347(+), B=4233, L=4061, M=4347, H=4486 | 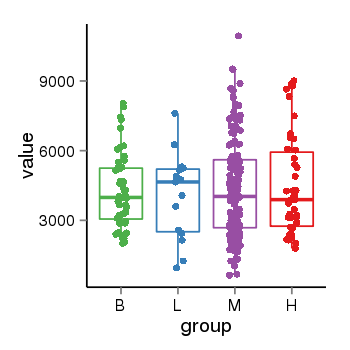 |
| 25399 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |