DMR 26458 chr2:128396051-128396350 (300bp)
| DMR Stats | |
|---|---|
| Position | chr2:128396051-128396350 (View on UCSC) |
| Width | 300bp |
| Genomic Context | 3' end |
| Nearest Genes | LIMS2 (0bp away) |
| ANODEV p-value | 0.035577779243 |
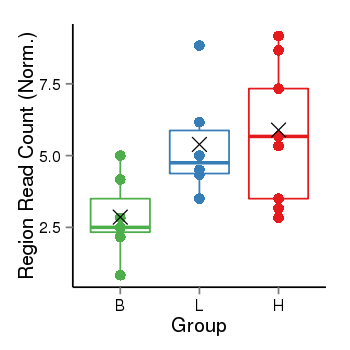
| Frequencies | Benign: 2.0 (28.57%), Low: 6.0 (100.0%), High: 8.0 (88.89 %) |
| Frequent? |
Region Count Boxplot
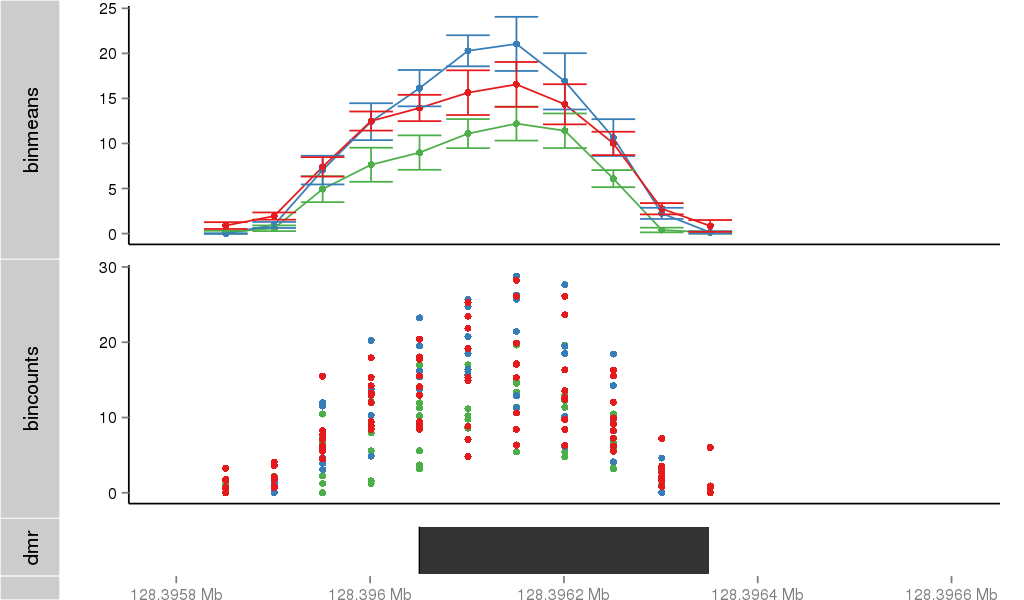
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 26458 | LIMS2 | 16955 | uc002tov.3 | chr2 | 128395996 | 128401685 | - | 300 | 100.0 | 0.16 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002tow.4 | chr2 | 128395996 | 128405828 | - | 300 | 100.0 | 0.12 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002tox.3 | chr2 | 128395996 | 128422167 | - | 300 | 100.0 | 0.31 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002toy.3 | chr2 | 128395996 | 128431883 | - | 300 | 100.0 | 0.23 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002toz.3 | chr2 | 128395996 | 128432764 | - | 300 | 100.0 | 0.12 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002tpa.3 | chr2 | 128395996 | 128432764 | - | 300 | 100.0 | 0.47 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc002tpb.3 | chr2 | 128395996 | 128439360 | - | 300 | 100.0 | 0.23 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc010fmb.3 | chr2 | 128395996 | 128422167 | - | 300 | 100.0 | 0.31 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (100) | 100.0 |
| 26458 | LIMS2 | 16955 | uc010yzm.2 | chr2 | 128395996 | 128432764 | - | 300 | 100.0 | 0.16 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (100) | 100.0 |
- 9 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 26458 | chr2 | 128395286 | 128396810 | wgEncodeRegDnaseClusteredV2 | 117 | score:1000, srow:650031 |
| 26458 | chr2 | 128395114 | 128396256 | wgEncodeRegTfbsClusteredV3 | POLR2A | score:550, expCount:8, expNums:22,78,163,206,207,256,525,619, expScores:121,234,319,515,368,241,159,131, srow:2279288 |
| 26458 | chr2 | 128395873 | 128396196 | wgEncodeRegTfbsClusteredV3 | CEBPB | score:363, expCount:7, expNums:172,212,343,425,426,462,477, expScores:141,207,157,179,347,204,363, srow:2279302 |
| 26458 | chr2 | 128395879 | 128396268 | wgEncodeRegTfbsClusteredV3 | MAX | score:242, expCount:2, expNums:223,511, expScores:242,184, srow:2279303 |
| 26458 | chr2 | 128395913 | 128396236 | wgEncodeRegTfbsClusteredV3 | E2F6 | score:206, expCount:1, expNums:215, expScores:206, srow:2279304 |
| 26458 | chr2 | 128395965 | 128396334 | wgEncodeRegTfbsClusteredV3 | MAZ | score:190, expCount:1, expNums:512, expScores:190, srow:2279305 |
| 26458 | chr2 | 128396303 | 128396313 | tfbsConsSites | V$AP1_Q2 | score:922, zScore:2.16, srow:2750491 |
| 26458 | chr2 | 128396087 | 128396089 | phastConsElements100way | lod=12 | score:240, srow:4764665 |
| 26458 | chr2 | 128396167 | 128396167 | gwasCatalog | rs78022502 | pubMedID:23535033, author:Sherva R, pubDate:2013-03-24, journal:Alzheimers Dement, title:Genome-wide association study of the rate of cognitive decline in Alzheimer's disease., trait:Alzheimer's disease (cognitive decline), initSample:303 European ancestry cases, replSample:, region:2q14.3, genes:LIMS2, riskAllele:rs78022502-?, riskAlFreq:0.06, pValue:2E-6, pValueDesc:, orOrBeta:.23, ci95:unit decrease, platform:Illumina [NR] (Imputed), cnv:N, srow:8931 |
- 9 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 26458 | cellmeth_recounts | PrEC: 40, LNCaP: 5, DU145: 3 | 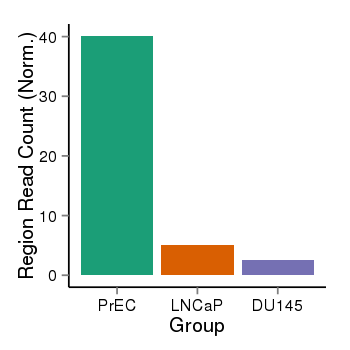 |
| 26458 | cellmeth_diffreps_PrECvsLNCaP | A=63, B=4, Length=520, Event=Down, log2FC=-3.98, padj=2.90251061721056e-13 | 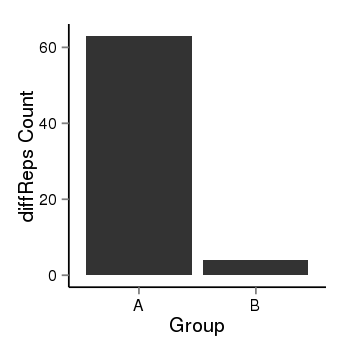 |
| 26458 | cellmeth_diffreps_PrECvsDU145 | A=63, B=4, Length=500, Event=Down, log2FC=-3.98, padj=3.96871667498526e-13 | 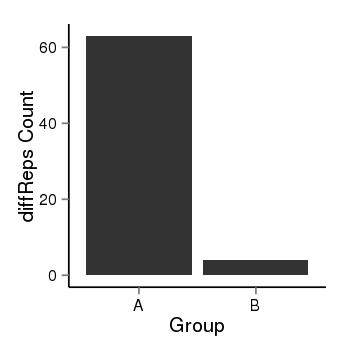 |
| 26458 | cellexp | id=ENSG00000072163, name=LIMS2, PrEC=2, str=69, LNCaP=22, str=11, DU145=229, str=182 | 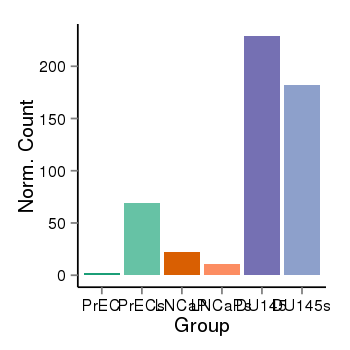 |
| 26458 | tcgaexp | gene=LIMS2, entrez=55679, pos=chr2:128395996-128439360(-), B=6847, L=6079, M=3186, H=2386 | 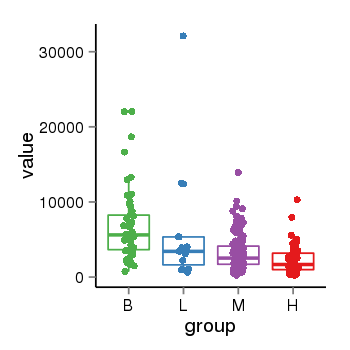 |