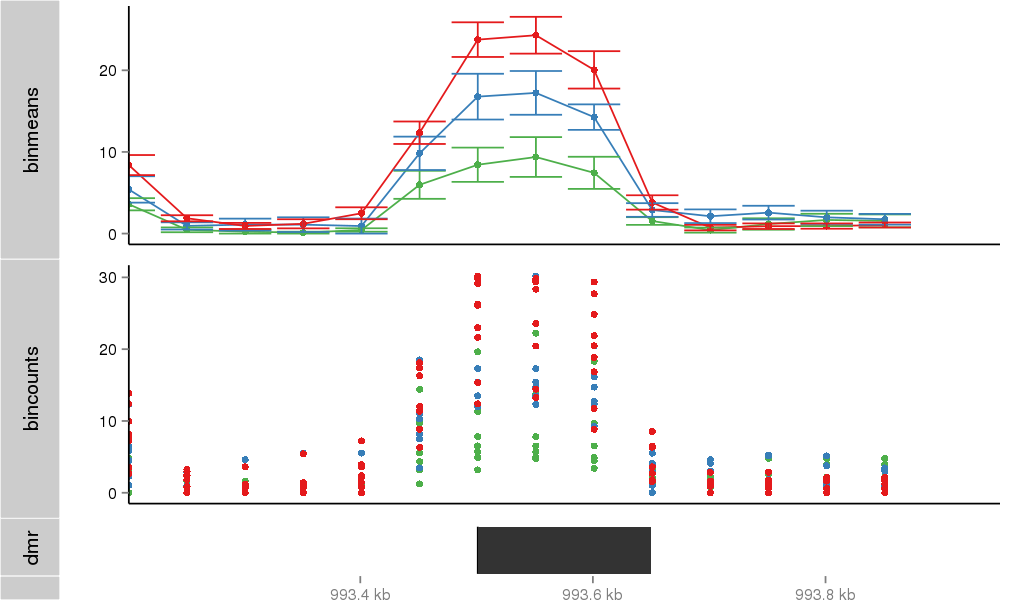
DMR 33895 chr17:993501-993650 (150bp)
| DMR Stats | |
|---|---|
| Position | chr17:993501-993650 (View on UCSC) |
| Width | 150bp |
| Genomic Context | intron |
| Nearest Genes | ABR (0bp away) |
| ANODEV p-value | 7.02415793855e-05 |
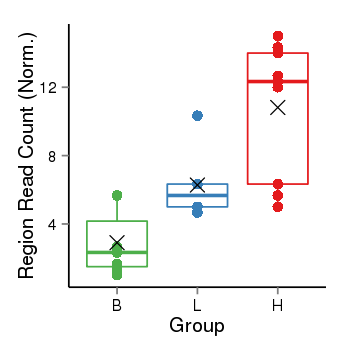
| Frequencies | Benign: 2.0 (28.57%), Low: 6.0 (100.0%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 33895 | ABR | 11892 | uc002fsd.4 | chr17 | 906758 | 1083166 | - | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (100), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0) | 0.0 |
| 33895 | ABR | 11892 | uc002fse.4 | chr17 | 906758 | 1090616 | - | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (100), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0) | 0.0 |
| 33895 | ABR | 11892 | uc002fsg.4 | chr17 | 906758 | 1012340 | - | 150 | 100.0 | 0.05 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (100), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |
| 33895 | ABR | 11892 | uc002fsh.1 | chr17 | 913969 | 995100 | - | 150 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (100), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0) | 0.0 |
- 4 geness
Feature Overlaps
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 33895 | cellmeth_recounts | PrEC: 1, LNCaP: 3, DU145: 1 | 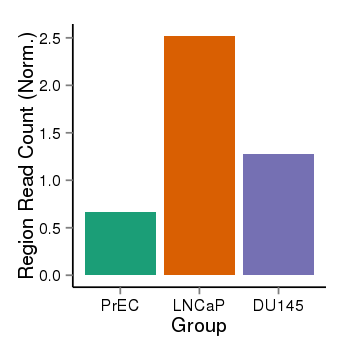 |
| 33895 | cellexp | id=ENSG00000159842, name=ABR, PrEC=5958, str=5449, LNCaP=7075, str=4383, DU145=7236, str=5886 | 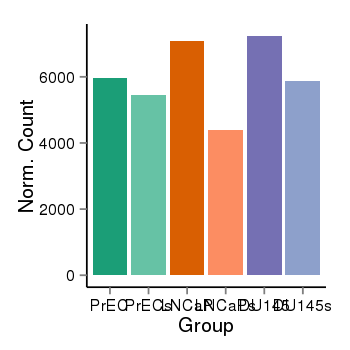 |
| 33895 | tcgaexp | gene=ABR, entrez=29, pos=chr17:906758-1090616(-), B=5740, L=5245, M=4792, H=4366 | 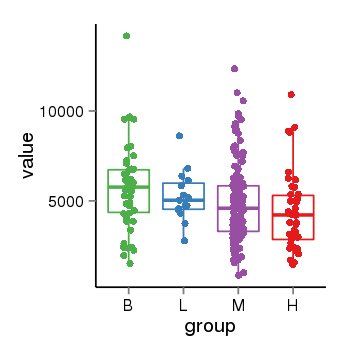 |
| 33895 | tcgaexp | gene=CRHR1, entrez=1394, pos=chr17:954315-43913194(+), B=36, L=21, M=25, H=18 | 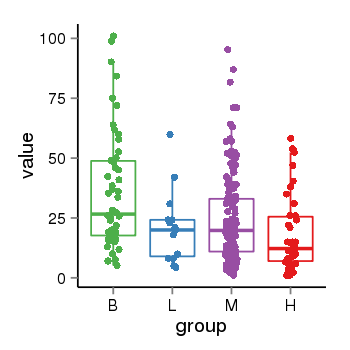 |
| 33895 | tcgaexp | gene=MAPT, entrez=4137, pos=chr17:762281-44105699(+), B=1933, L=2382, M=2487, H=2583 | 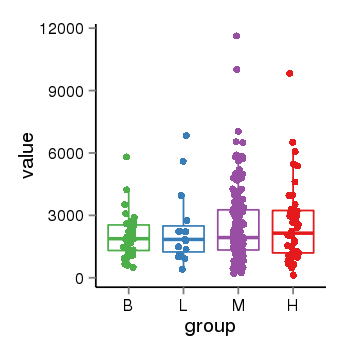 |
| 33895 | tcgaexp | gene=NSF, entrez=4905, pos=chr17:266212-44834828(+), B=3355, L=5678, M=5410, H=5700 | 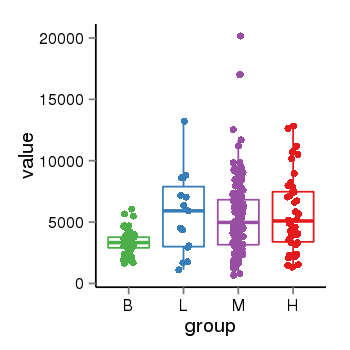 |
| 33895 | tcgaexp | gene=PLEKHM1, entrez=9842, pos=chr17:128328-43568146(-), B=1993, L=1631, M=1589, H=1418 | 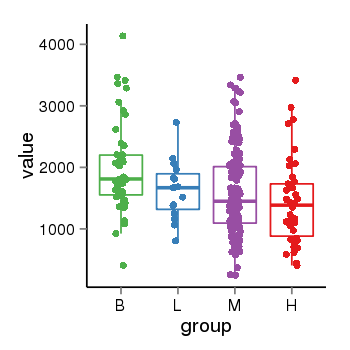 |
| 33895 | tcgaexp | gene=LRRC37A, entrez=9884, pos=chr17:415627-44415160(+), B=924, L=896, M=971, H=940 | 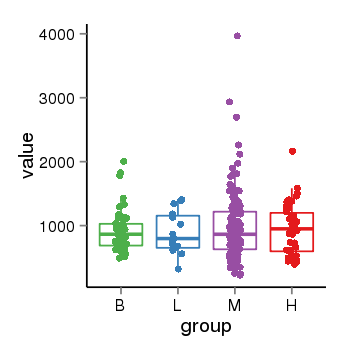 |
| 33895 | tcgaexp | gene=ARL17A, entrez=51326, pos=chr17:195721-44657088(-), B=200, L=195, M=210, H=230 | 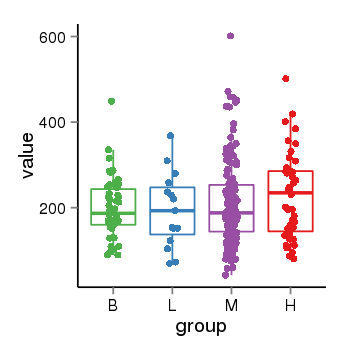 |
| 33895 | tcgaexp | gene=LRRC37A4P, entrez=55073, pos=chr17:197706-43597889(-), B=1015, L=1512, M=1396, H=1450 | 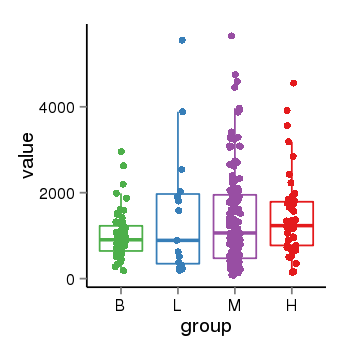 |
| 33895 | tcgaexp | gene=SPPL2C, entrez=162540, pos=chr17:943102-43924438(+), B=0, L=0, M=0, H=0 | 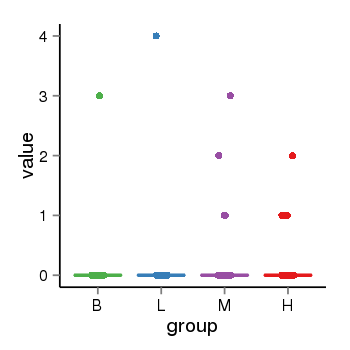 |
| 33895 | tcgaexp | gene=ARHGAP27, entrez=201176, pos=chr17:86404-43511112(-), B=1570, L=1264, M=1273, H=1486 | 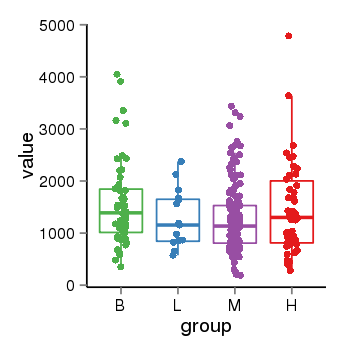 |
| 33895 | tcgaexp | gene=STH, entrez=246744, pos=chr17:790689-44077060(+), B=0, L=0, M=0, H=0 | 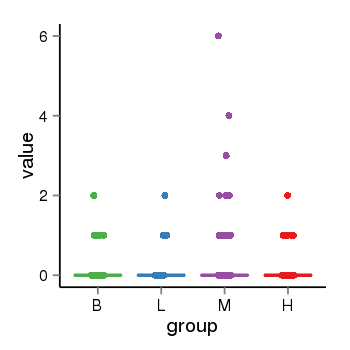 |
| 33895 | tcgaexp | gene=KANSL1, entrez=284058, pos=chr17:563498-44302740(-), B=3141, L=2594, M=2797, H=3017 | 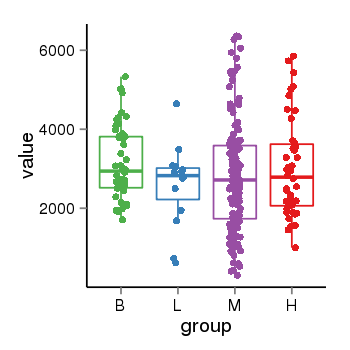 |
| 33895 | tcgaexp | gene=LRRC37A2, entrez=474170, pos=chr17:197706-44633014(+), B=351, L=310, M=361, H=377 | 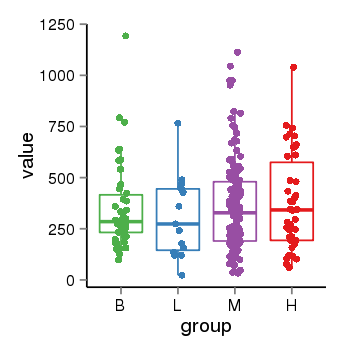 |
| 33895 | tcgaexp | gene=MAPT-AS1, entrez=100128977, pos=chr17:894694-43972879(-), B=1, L=2, M=4, H=2 | 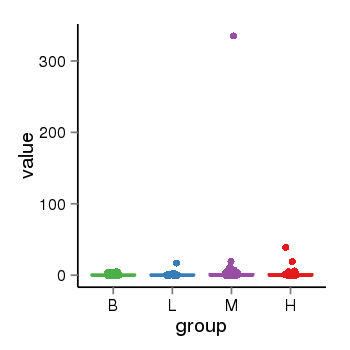 |
| 33895 | tcgaexp | gene=MAPT-IT1, entrez=100130148, pos=chr17:891434-43976164(+), B=5, L=3, M=3, H=4 | 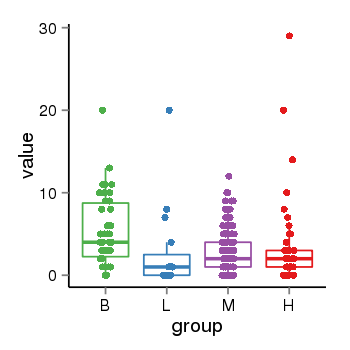 |