DMR 36 chr1:3147701-3147900 (200bp)
| DMR Stats | |
|---|---|
| Position | chr1:3147701-3147900 (View on UCSC) |
| Width | 200bp |
| Genomic Context | intron |
| Nearest Genes | PRDM16 (0bp away) |
| ANODEV p-value | 0.00115276908657 |
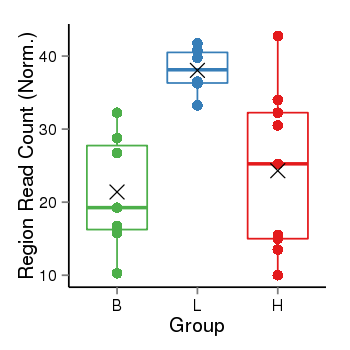
| Frequencies | Benign: 7.0 (100.0%), Low: 6.0 (100.0%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot
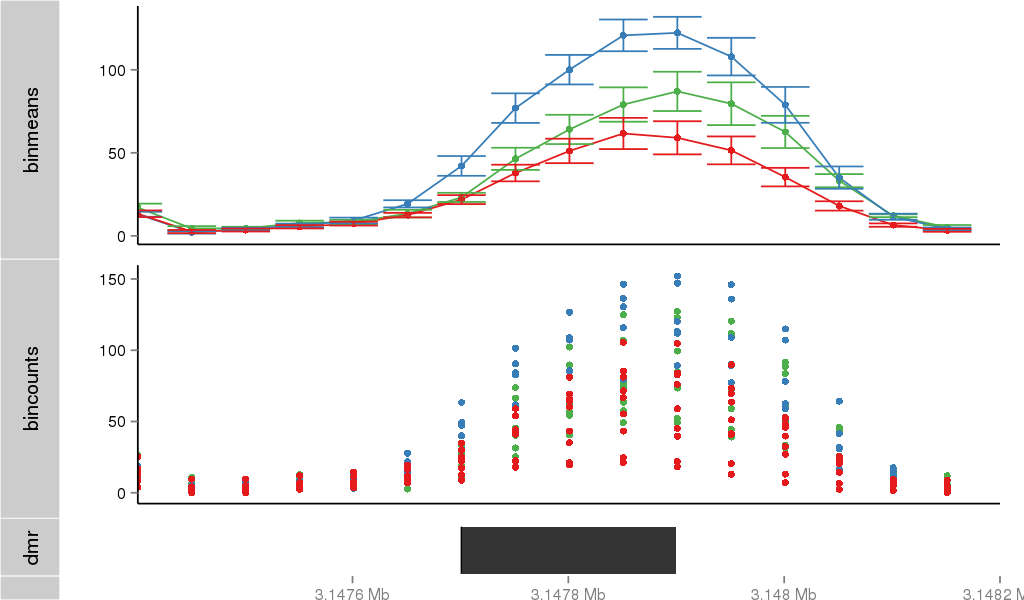
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 36 | PRDM16 | 117 | uc001akc.3 | chr1 | 2985742 | 3355185 | + | 200 | 100.0 | 1.51 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 36 | PRDM16 | 117 | uc001ake.3 | chr1 | 2985742 | 3355185 | + | 200 | 100.0 | 1.51 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 36 | PRDM16 | 117 | uc001akf.3 | chr1 | 2985742 | 3355185 | + | 200 | 100.0 | 1.51 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 36 | PRDM16 | 117 | uc009vlh.3 | chr1 | 2985742 | 3355185 | + | 200 | 100.0 | 1.52 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
- 4 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 36 | chr1 | 3147846 | 3148081 | cpgIsland | CpG: 19 | length:236, cpgNum:19, gcNum:149, perCpg:16.1, perGc:63.1, obsExp:0.81, srow:248 |
| 36 | chr1 | 3145846 | 3147845 | cpgShore | — | values(x.gr)[overs$srow, ]:403 |
- 2 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 36 | cellmeth_recounts | PrEC: 112, LNCaP: 0, DU145: 12 | 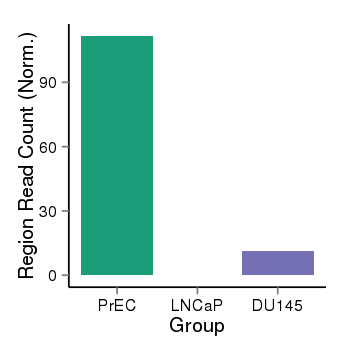 |
| 36 | cellmeth_diffreps_PrECvsLNCaP | A=297, B=1, Length=1100, Event=Down, log2FC=-8.21, padj=0 | 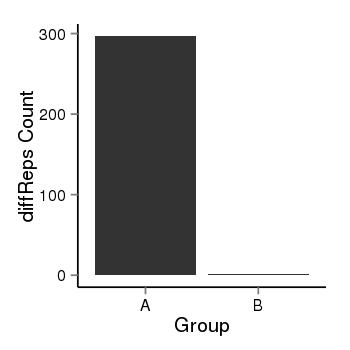 |
| 36 | cellmeth_diffreps_PrECvsDU145 | A=297, B=14, Length=1080, Event=Down, log2FC=-4.41, padj=0 | 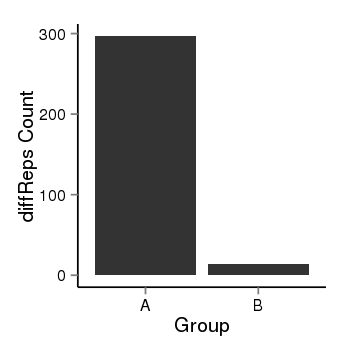 |
| 36 | cellexp | id=ENSG00000142611, name=PRDM16, PrEC=8, str=18, LNCaP=18, str=10, DU145=49, str=42 | 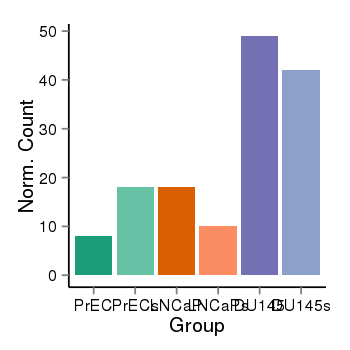 |
| 36 | tcgaexp | gene=SRSF10, entrez=10772, pos=chr1:36273-24306953(-), B=3487, L=3388, M=3432, H=4019 | 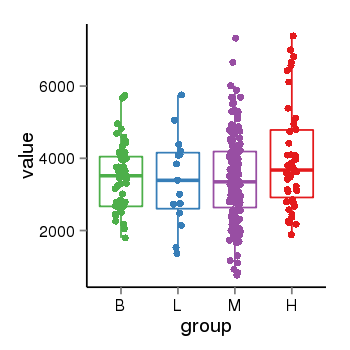 |
| 36 | tcgaexp | gene=PRDM16, entrez=63976, pos=chr1:2985742-3355185(+), B=306, L=40, M=53, H=56 | 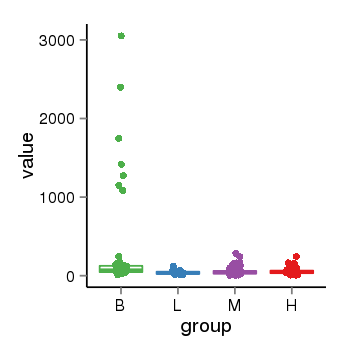 |
| 36 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |