DMR 37387 chr2:240212651-240212950 (300bp)
| DMR Stats | |
|---|---|
| Position | chr2:240212651-240212950 (View on UCSC) |
| Width | 300bp |
| Genomic Context | intron |
| Nearest Genes | HDAC4 (0bp away) |
| ANODEV p-value | 0.000348795862278 |
| Frequencies | Benign: 1.0 (14.29%), Low: 2.0 (33.33%), High: 8.0 (88.89 %) |
| Frequent? |
Region Count Boxplot

Region Overview

Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 37387 | HDAC4 | 17807 | uc002vyk.4 | chr2 | 239969864 | 240322643 | - | 300 | 100.0 | 1.61 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0) | 0.0 |
| 37387 | HDAC4 | 17807 | uc010fyz.1 | chr2 | 240011372 | 240220334 | - | 300 | 100.0 | 4.42 | 0 | 0.0 | E1 (0), I1 (100), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 37387 | HDAC4 | 17807 | uc010fza.2 | chr2 | 240016312 | 240323346 | - | 300 | 100.0 | 1.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 37387 | HDAC4 | 17807 | uc010zoa.1 | chr2 | 240016312 | 240220334 | - | 300 | 100.0 | 1.01 | 0 | 0.0 | E1 (0), I1 (100), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
- 4 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 37387 | chr2 | 240212486 | 240213155 | wgEncodeRegDnaseClusteredV2 | 45 | score:1000, srow:695996 |
| 37387 | chr2 | 240212084 | 240212713 | wgEncodeRegTfbsClusteredV3 | STAT5A | score:493, expCount:1, expNums:114, expScores:493, srow:2430645 |
| 37387 | chr2 | 240212098 | 240212693 | wgEncodeRegTfbsClusteredV3 | NFIC | score:277, expCount:1, expNums:97, expScores:277, srow:2430646 |
| 37387 | chr2 | 240212120 | 240212669 | wgEncodeRegTfbsClusteredV3 | POLR2A | score:298, expCount:1, expNums:130, expScores:298, srow:2430647 |
| 37387 | chr2 | 240212526 | 240212955 | wgEncodeRegTfbsClusteredV3 | CTCF | score:699, expCount:26, expNums:5,16,35,37,39,42,44,463,597,609,635,641,642,643,644,657,658,659,664,671,672,675,676,683,684,686, expScores:145,177,215,162,649,130,281,134,376,126,699,273,304,258,554,418,325,184,646,430,181,154,209,383,83,435, srow:2430654 |
| 37387 | chr2 | 240212630 | 240212875 | wgEncodeRegTfbsClusteredV3 | RAD21 | score:213, expCount:2, expNums:149,466, expScores:213,157, srow:2430655 |
- 6 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 37387 | cellmeth_recounts | PrEC: 0, LNCaP: 9, DU145: 8 |  |
| 37387 | cellexp | id=ENSG00000068024, name=HDAC4, PrEC=765, str=570, LNCaP=803, str=661, DU145=1779, str=1296 | 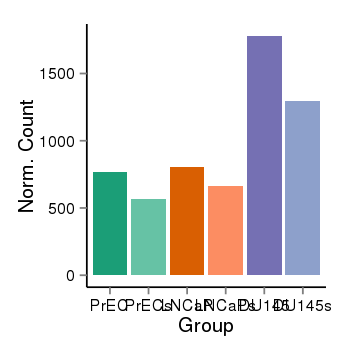 |
| 37387 | tcgameth | site:cg27168632, B=0.6, L=0.82, H=0.8 |  |
| 37387 | tcgaexp | gene=HDAC4, entrez=9759, pos=chr2:239969864-240323346(-), B=1448, L=1412, M=1302, H=1292 | 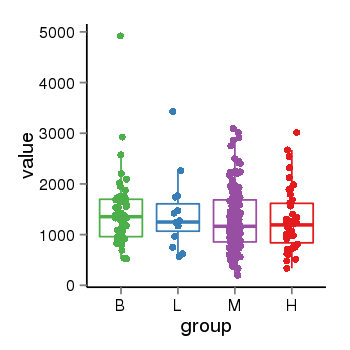 |
| 37387 | pubmed | gene=HDAC4, G=501, GP=12, GC=161, GM=27, GPM=1, GCM=11 |  |