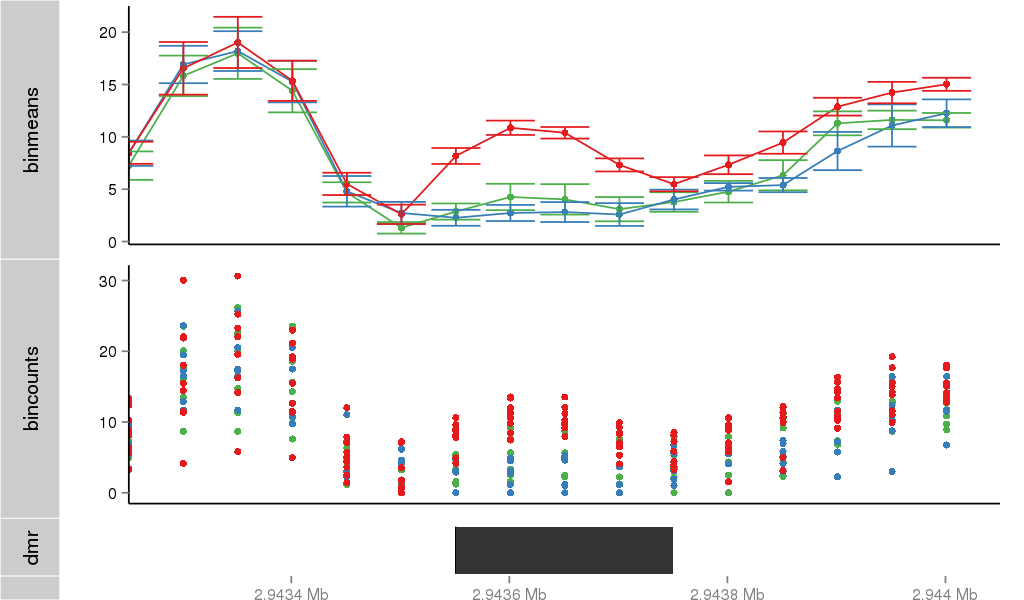
DMR 37758 chr4:2943551-2943750 (200bp)
| DMR Stats | |
|---|---|
| Position | chr4:2943551-2943750 (View on UCSC) |
| Width | 200bp |
| Genomic Context | 3' end |
| Nearest Genes | NOP14-AS1, AB000466, NOP14 (0bp away) |
| ANODEV p-value | 2.70016576378e-05 |
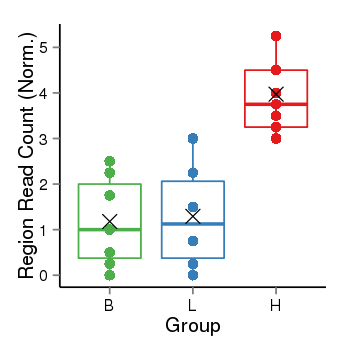
| Frequencies | Benign: 0.0 (0.0%), Low: 0.0 (0.0%), High: 8.0 (88.89 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 37758 | NOP14-AS1 | 21309 | uc003ggd.1 | chr4 | 2936626 | 2943596 | + | 46 | 23.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (23) | 100.0 |
| 37758 | NOP14-AS1 | 21309 | uc003gge.1 | chr4 | 2936626 | 2949400 | + | 200 | 100.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (100), E5 (0) | 0.0 |
| 37758 | AB000466 | 21309 | uc003ggg.1 | chr4 | 2937278 | 2943596 | + | 46 | 23.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (23) | 100.0 |
| 37758 | NOP14-AS1 | 21309 | uc003ggh.3 | chr4 | 2937278 | 2952794 | + | 200 | 100.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0) | 0.0 |
| 37758 | NOP14-AS1 | 21309 | uc003ggi.1 | chr4 | 2938993 | 2963465 | + | 200 | 100.0 | 0.04 | 1 | 0.0 | E1 (0), I1 (100), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0) | 0.0 |
| 37758 | NOP14 | 21310 | uc003ggj.1 | chr4 | 2939664 | 2965118 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (100), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0) | 0.0 |
| 37758 | NOP14 | 21310 | uc003ggl.3 | chr4 | 2940325 | 2965118 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (100), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0) | 0.0 |
| 37758 | NOP14 | 21310 | uc010icp.2 | chr4 | 2939664 | 2955372 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (100), E11 (0) | 0.0 |
- 8 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 37758 | chr4 | 2942156 | 2945442 | wgEncodeRegTfbsClusteredV3 | POLR2A | score:683, expCount:23, expNums:61,62,105,123,124,130,131,146,147,168,306,334,340,402,403,522,527,564,577,602,622,632,634, expScores:683,502,458,282,319,332,545,257,286,295,330,152,222,320,246,175,178,168,159,150,222,384,207, srow:2995734 |
| 37758 | chr4 | 2943734 | 2944003 | wgEncodeRegTfbsClusteredV3 | CTCF | score:147, expCount:2, expNums:5,137, expScores:121,147, srow:2995736 |
- 2 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 37758 | cellmeth_recounts | PrEC: 13, LNCaP: 3, DU145: 4 | 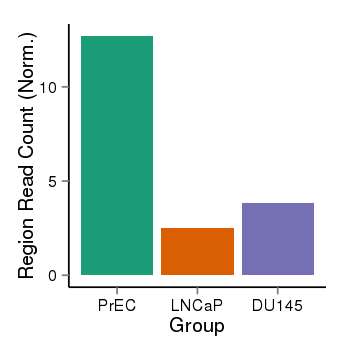 |
| 37758 | cellmeth_diffreps_PrECvsLNCaP | A=74, B=7, Length=800, Event=Down, log2FC=-3.4, padj=1.03552333609664e-13 | 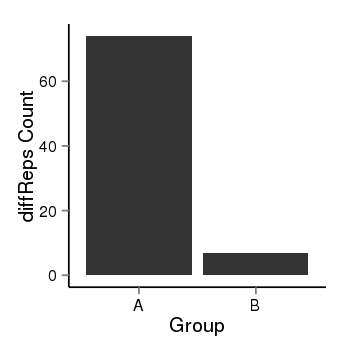 |
| 37758 | cellmeth_diffreps_PrECvsDU145 | A=121, B=8, Length=1320, Event=Down, log2FC=-3.92, padj=0 | 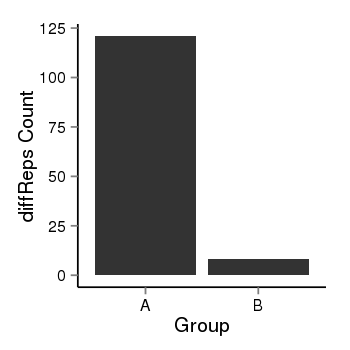 |
| 37758 | cellexp | id=ENSG00000087269, name=NOP14, PrEC=4886, str=2415, LNCaP=4228, str=4865, DU145=3836, str=4571 | 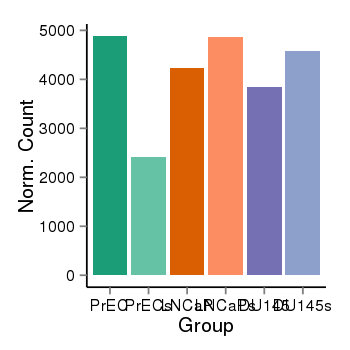 |
| 37758 | cellexp | id=ENSG00000249673, name=NOP14-AS1, PrEC=2120, str=2870, LNCaP=1244, str=978, DU145=2660, str=3168 | 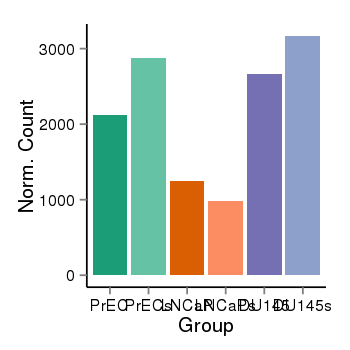 |
| 37758 | tcgaexp | gene=UGT2B10, entrez=7365, pos=chr4:393964-69886113(+), B=12, L=1, M=2, H=8 | 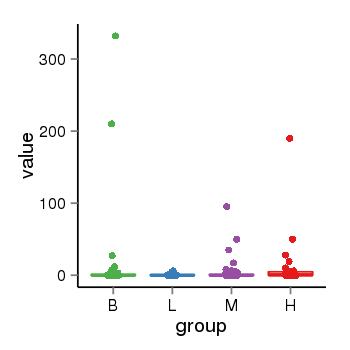 |
| 37758 | tcgaexp | gene=NOP14, entrez=8602, pos=chr4:2939664-2965118(-), B=2099, L=2792, M=2677, H=2758 | 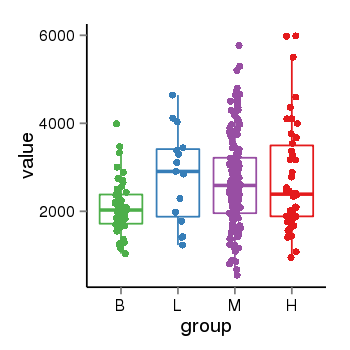 |
| 37758 | tcgaexp | gene=UGT2A3, entrez=79799, pos=chr4:506428-69817509(-), B=1, L=0, M=1, H=1 | 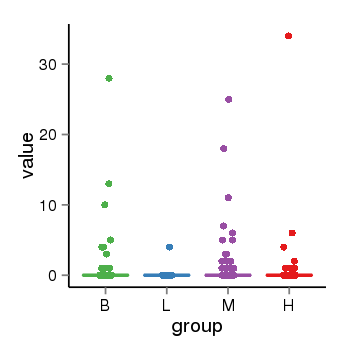 |
| 37758 | tcgaexp | gene=YTHDC1, entrez=91746, pos=chr4:6027-69215824(-), B=4512, L=3976, M=4109, H=4458 | 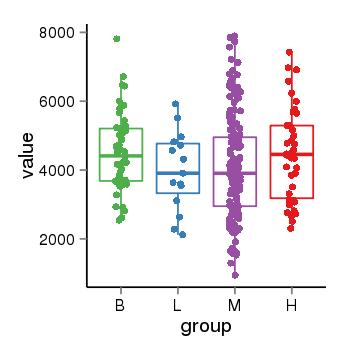 |
| 37758 | tcgaexp | gene=NOP14-AS1, entrez=317648, pos=chr4:2936626-2963465(+), B=971, L=768, M=808, H=775 | 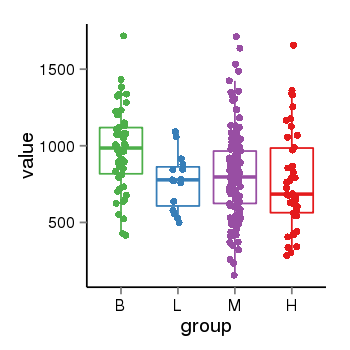 |
| 37758 | tcgaexp | gene=DUX4L5, entrez=653545, pos=chr4:72836-191010142(+), B=0, L=0, M=0, H=0 | 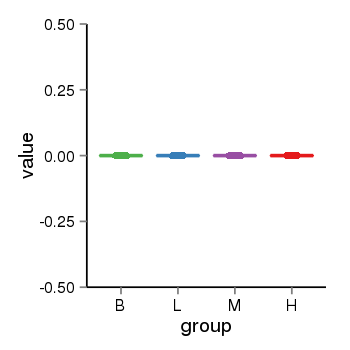 |
| 37758 | pubmed | gene=NOP14-AS1, G=0, GP=0, GC=0, GM=0, GPM=0, GCM=0 | 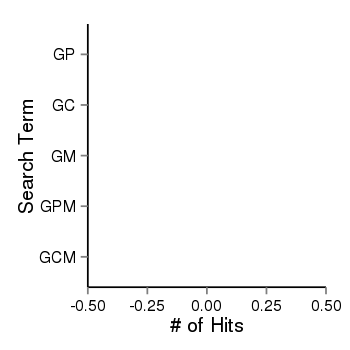 |
| 37758 | pubmed | gene=NOP14, G=10, GP=0, GC=3, GM=0, GPM=0, GCM=0 | 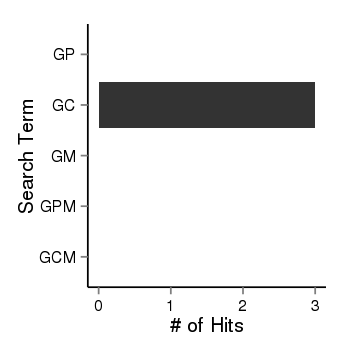 |