DMR 39772 chr10:126692051-126692450 (400bp)
| DMR Stats | |
|---|---|
| Position | chr10:126692051-126692450 (View on UCSC) |
| Width | 400bp |
| Genomic Context | exon |
| Nearest Genes | CTBP2 (0bp away) |
| ANODEV p-value | 0.0111192193578 |
| Frequencies | Benign: 0.0 (0.0%), Low: 2.0 (33.33%), High: 7.0 (77.78 %) |
| Frequent? |
Region Count Boxplot

Region Overview

Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 39772 | CTBP2 | 3988 | uc001lid.4 | chr10 | 126676418 | 126694582 | - | 400 | 100.0 | 1.63 | 0 | 0.0 | E1 (0), I1 (97.5), E2 (2.75), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0) | 0.0 |
| 39772 | CTBP2 | 3988 | uc001lie.4 | chr10 | 126676418 | 126716453 | - | 400 | 100.0 | 2.04 | 0 | 0.0 | E1 (0), I1 (97.5), E2 (2.75), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0) | 0.0 |
| 39772 | CTBP2 | 3988 | uc001lif.4 | chr10 | 126676418 | 126849103 | - | 400 | 100.0 | 1.07 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (97.5), E4 (2.75), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0) | 0.0 |
| 39772 | CTBP2 | 3988 | uc001lih.4 | chr10 | 126676418 | 126849624 | - | 400 | 100.0 | 0.81 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (97.5), E4 (2.75), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0) | 0.0 |
| 39772 | CTBP2 | 3988 | uc009yak.3 | chr10 | 126676418 | 126847285 | - | 400 | 100.0 | 1.88 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (97.5), E4 (2.75), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0) | 0.0 |
| 39772 | CTBP2 | 3988 | uc009yal.3 | chr10 | 126676418 | 126849103 | - | 400 | 100.0 | 0.53 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (97.5), E4 (2.75), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0) | 0.0 |
- 6 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 39772 | chr10 | 126691826 | 126692655 | wgEncodeRegDnaseClusteredV2 | 28 | score:447, srow:178116 |
| 39772 | chr10 | 126691781 | 126692210 | wgEncodeRegTfbsClusteredV3 | CTCF | score:281, expCount:25, expNums:5,44,595,597,603,607,609,627,628,630,631,646,648,652,655,659,660,662,663,665,667,669,678,685,688, expScores:126,131,281,119,150,121,152,199,186,251,207,141,109,137,100,229,101,147,134,130,115,141,115,118,124, srow:628257 |
| 39772 | chr10 | 126692052 | 126692065 | tfbsConsSites | V$GATA1_03 | score:885, zScore:2.45, srow:742996 |
| 39772 | chr10 | 126692054 | 126692063 | tfbsConsSites | V$GATA2_01 | score:946, zScore:1.83, srow:742997 |
| 39772 | chr10 | 126692025 | 126692064 | phastConsElements100way | lod=519 | score:612, srow:1286253 |
- 5 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 39772 | cellmeth_recounts | PrEC: 31, LNCaP: 10, DU145: 9 |  |
| 39772 | cellmeth_diffreps_PrECvsLNCaP | A=34, B=5, Length=340, Event=Down, log2FC=-2.77, padj=4.44240505496598e-05 |  |
| 39772 | cellmeth_diffreps_PrECvsDU145 | A=36, B=7, Length=380, Event=Down, log2FC=-2.36, padj=0.000210273291496912 |  |
| 39772 | cellexp | id=ENSG00000175029, name=CTBP2, PrEC=7973, str=5485, LNCaP=6592, str=5922, DU145=8112, str=7309 |  |
| 39772 | tcgaexp | gene=CTBP2, entrez=1488, pos=chr10:126676418-126849624(-), B=5593, L=5696, M=6014, H=6079 |  |
| 39772 | tcgaexp | gene=DUX4, entrez=22947, pos=chr10:76142-191000255(+), B=0, L=0, M=0, H=0 | 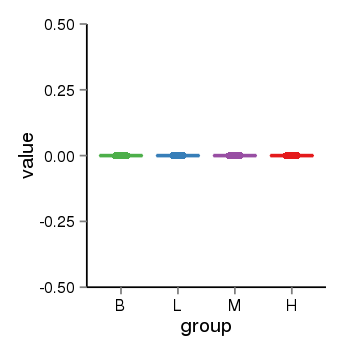 |
| 39772 | tcgaexp | gene=FAM45B, entrez=55855, pos=chr10:120863611-129631421(+), B=500, L=423, M=440, H=462 |  |