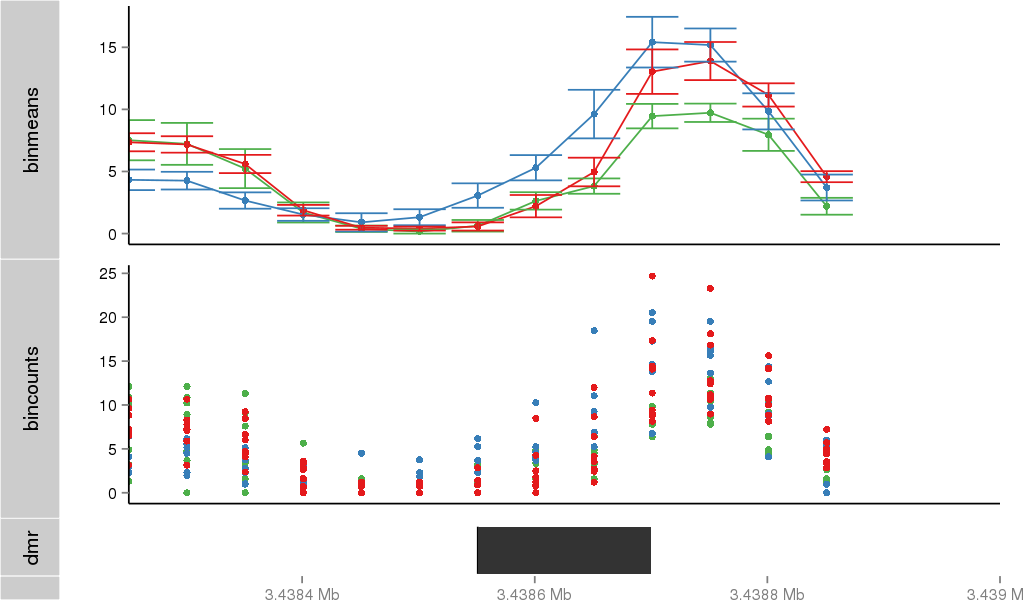
DMR 40 chr1:3438551-3438700 (150bp)
| DMR Stats | |
|---|---|
| Position | chr1:3438551-3438700 (View on UCSC) |
| Width | 150bp |
| Genomic Context | intron |
| Nearest Genes | MEGF6 (0bp away) |
| ANODEV p-value | 0.012189774093 |
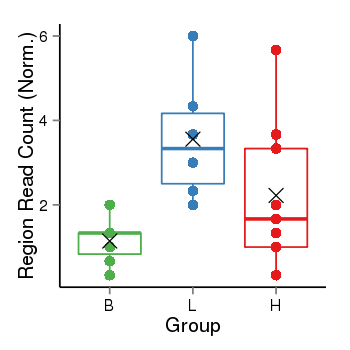
| Frequencies | Benign: 0.0 (0.0%), Low: 3.0 (50.0%), High: 3.0 (33.33 %) |
| Frequent? |
Region Count Boxplot
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 40 | MEGF6 | 120 | uc001akk.3 | chr1 | 3404506 | 3448012 | - | 150 | 100.0 | 1.14 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0), I27 (0), E28 (0), I28 (0), E29 (0), I29 (0), E30 (0) | 0.0 |
| 40 | MEGF6 | 120 | uc001akl.3 | chr1 | 3404506 | 3528059 | - | 150 | 100.0 | 1.53 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (100), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0), I27 (0), E28 (0), I28 (0), E29 (0), I29 (0), E30 (0), I30 (0), E31 (0), I31 (0), E32 (0), I32 (0), E33 (0), I33 (0), E34 (0), I34 (0), E35 (0), I35 (0), E36 (0), I36 (0), E37 (0) | 0.0 |
- 2 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 40 | chr1 | 3438251 | 3440250 | cpgShelf | — | values(x.gr)[overs$srow, ]:281 |
- 1 features
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 40 | cellmeth_recounts | PrEC: 11, LNCaP: 0, DU145: 1 | 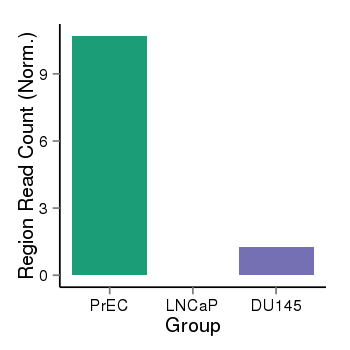 |
| 40 | cellmeth_diffreps_PrECvsLNCaP | A=37, B=2, Length=400, Event=Down, log2FC=-4.21, padj=4.52212701380725e-08 | 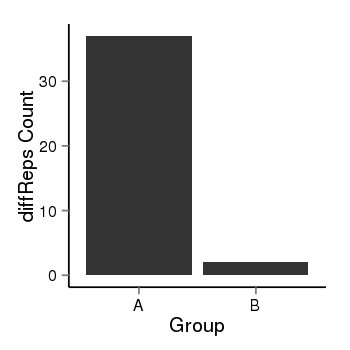 |
| 40 | cellmeth_diffreps_PrECvsDU145 | A=37, B=1, Length=420, Event=Down, log2FC=-5.21, padj=4.91555057723503e-09 | 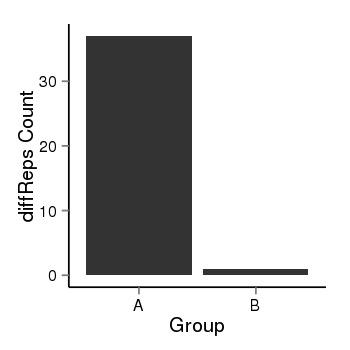 |
| 40 | cellexp | id=ENSG00000162591, name=MEGF6, PrEC=207, str=606, LNCaP=365, str=127, DU145=1319, str=853 | 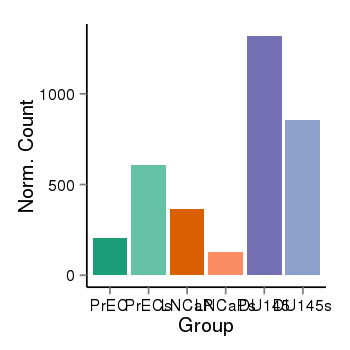 |
| 40 | tcgaexp | gene=MEGF6, entrez=1953, pos=chr1:3404506-3528059(-), B=2248, L=2404, M=2137, H=1929 | 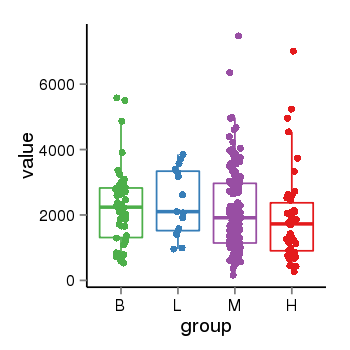 |
| 40 | tcgaexp | gene=SRSF10, entrez=10772, pos=chr1:36273-24306953(-), B=3487, L=3388, M=3432, H=4019 | 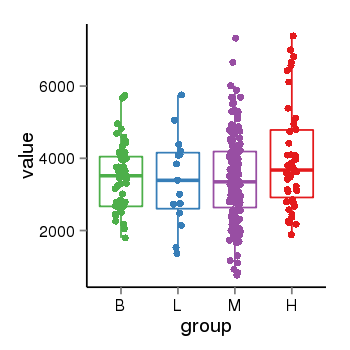 |
| 40 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |