DMR 41084 chr17:468901-469050 (150bp)
| DMR Stats | |
|---|---|
| Position | chr17:468901-469050 (View on UCSC) |
| Width | 150bp |
| Genomic Context | intron |
| Nearest Genes | VPS53 (0bp away) |
| ANODEV p-value | 0.000405861948565 |
| Frequencies | Benign: 2.0 (28.57%), Low: 2.0 (33.33%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot

Region Overview

Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 41084 | VPS53 | 11881 | uc002frk.3 | chr17 | 411908 | 579647 | - | 150 | 100.0 | 0.9 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (100), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc002frl.2 | chr17 | 429209 | 618096 | - | 150 | 100.0 | 0.13 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (100), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc002frm.2 | chr17 | 435533 | 618096 | - | 150 | 100.0 | 0.56 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (100), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc002frn.2 | chr17 | 435533 | 618096 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (100), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc002fro.2 | chr17 | 435533 | 618096 | - | 150 | 100.0 | 0.37 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (100), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc010cjo.2 | chr17 | 411908 | 618096 | - | 150 | 100.0 | 0.06 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (100), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0), I17 (0), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0) | 0.0 |
| 41084 | VPS53 | 11881 | uc010cjp.1 | chr17 | 435533 | 618096 | - | 150 | 100.0 | 0.06 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (100), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0) | 0.0 |
- 7 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 41084 | chr17 | 468806 | 469035 | wgEncodeRegDnaseClusteredV2 | 17 | score:336, srow:471256 |
| 41084 | chr17 | 468709 | 469175 | wgEncodeRegTfbsClusteredV3 | CTCF | score:1000, expCount:72, expNums:5,9,12,16,18,20,24,37,39,42,44,48,49,137,174,213,262,267,463,493,595,597,598,603,609,612,615,621,627,628,629,630,631,636,638,639,640,641,642,645,647,652,655,657,658,659,661,662,663,665,666,667,668,669,671,672,673,674,675,676,677,678,679,680,681,682,683,685,686,687,688,689, expScores:356,511,424,340,238,255,290,222,158,223,296,374,352,941,1000,1000,135,172,232,483,941,218,462,147,238,218,132,209,768,1000,1000,766,1000,164,685,181,152,127,186,230,294,153,102,117,129,686,138,426,500,436,324,534,106,345,206,159,356,163,242,230,344,242,220,569,103,135,169,477,154,282,444,92, srow:1606700 |
| 41084 | chr17 | 468792 | 469127 | wgEncodeRegTfbsClusteredV3 | POLR2A | score:187, expCount:1, expNums:256, expScores:187, srow:1606701 |
| 41084 | chr17 | 468824 | 469069 | wgEncodeRegTfbsClusteredV3 | RAD21 | score:229, expCount:2, expNums:149,193, expScores:225,229, srow:1606702 |
- 4 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 41084 | cellmeth_recounts | PrEC: 8, LNCaP: 4, DU145: 13 |  |
| 41084 | cellmeth_diffreps_PrECvsDU145 | A=2, B=32, Length=420, Event=Up, log2FC=4, padj=1.36954646948928e-06 |  |
| 41084 | cellmeth_diffreps_LNCaPvsDU145 | A=1.07, B=29.93, Length=460, Event=Up, log2FC=4.81, padj=1.86016333064862e-06 |  |
| 41084 | cellexp | id=ENSG00000141252, name=VPS53, PrEC=5002, str=7683, LNCaP=2639, str=3178, DU145=2836, str=2414 | 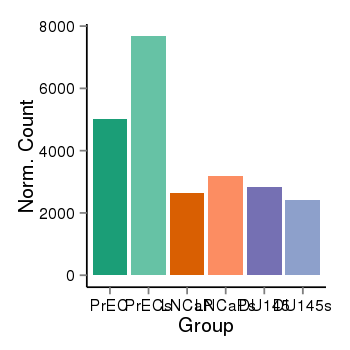 |
| 41084 | tcgaexp | gene=NSF, entrez=4905, pos=chr17:266212-44834828(+), B=3355, L=5678, M=5410, H=5700 | 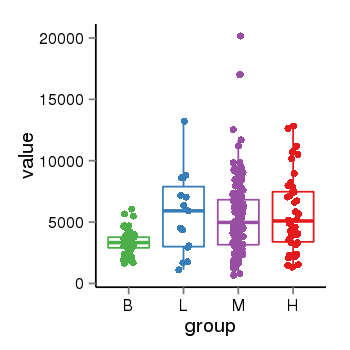 |
| 41084 | tcgaexp | gene=PLEKHM1, entrez=9842, pos=chr17:128328-43568146(-), B=1993, L=1631, M=1589, H=1418 | 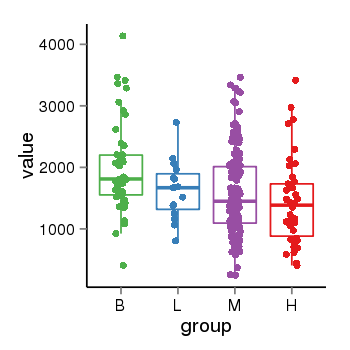 |
| 41084 | tcgaexp | gene=LRRC37A, entrez=9884, pos=chr17:415627-44415160(+), B=924, L=896, M=971, H=940 | 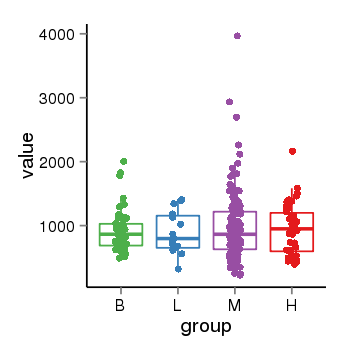 |
| 41084 | tcgaexp | gene=ARL17A, entrez=51326, pos=chr17:195721-44657088(-), B=200, L=195, M=210, H=230 | 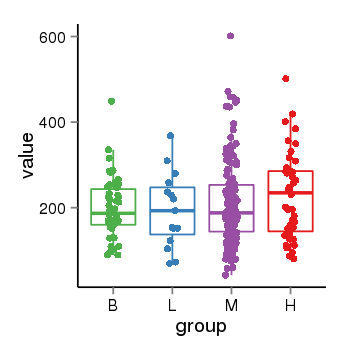 |
| 41084 | tcgaexp | gene=LRRC37A4P, entrez=55073, pos=chr17:197706-43597889(-), B=1015, L=1512, M=1396, H=1450 | 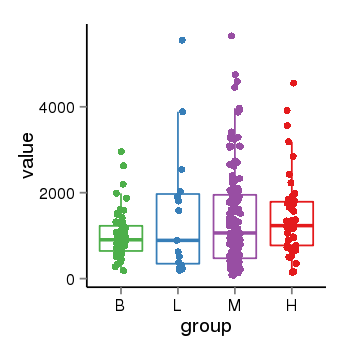 |
| 41084 | tcgaexp | gene=VPS53, entrez=55275, pos=chr17:411908-618096(-), B=2430, L=3151, M=2976, H=2590 | 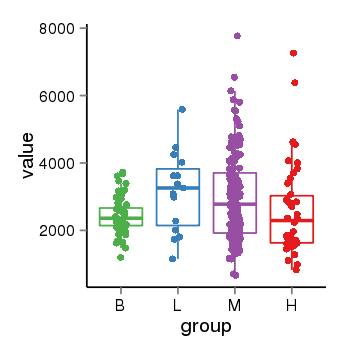 |
| 41084 | tcgaexp | gene=ARHGAP27, entrez=201176, pos=chr17:86404-43511112(-), B=1570, L=1264, M=1273, H=1486 | 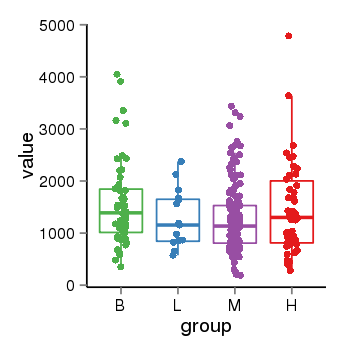 |
| 41084 | tcgaexp | gene=LRRC37A2, entrez=474170, pos=chr17:197706-44633014(+), B=351, L=310, M=361, H=377 | 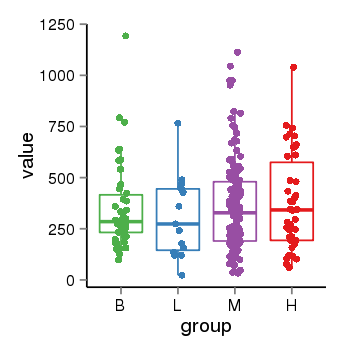 |
| 41084 | pubmed | gene=VPS53, G=30, GP=0, GC=10, GM=0, GPM=0, GCM=0 |  |