DMR 526 chr1:179068751-179068900 (150bp)
| DMR Stats | |
|---|---|
| Position | chr1:179068751-179068900 (View on UCSC) |
| Width | 150bp |
| Genomic Context | 3' end |
| Nearest Genes | ABL2 (0bp away) |
| ANODEV p-value | 0.0232163548303 |
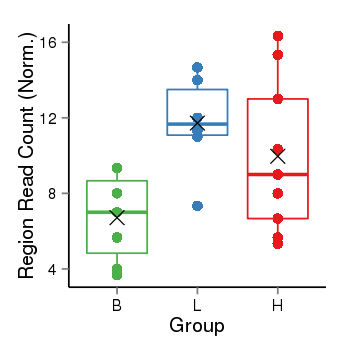
| Frequencies | Benign: 7.0 (100.0%), Low: 6.0 (100.0%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot
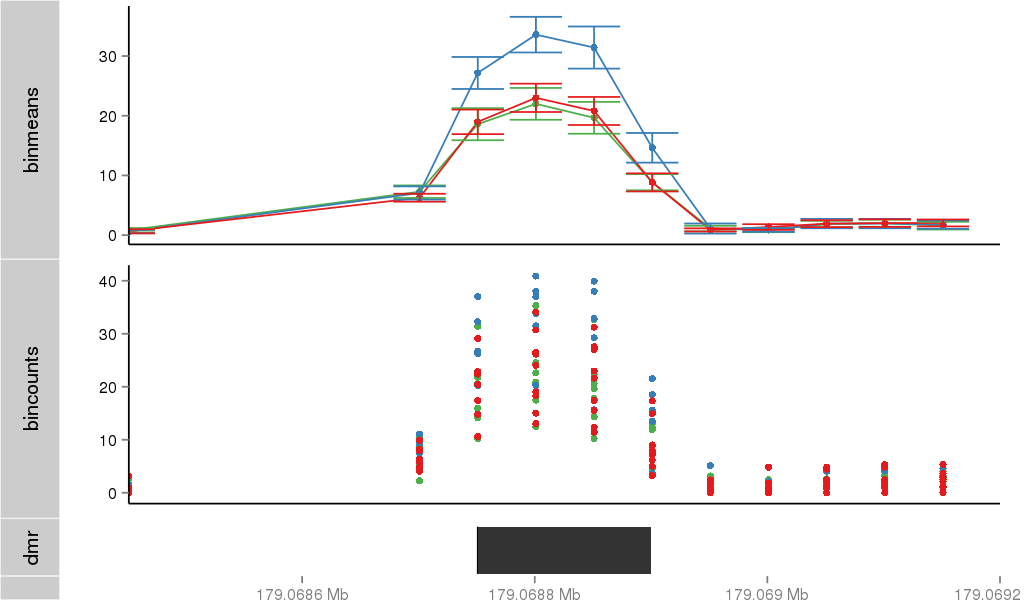
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 526 | ABL2 | 2212 | uc001gmg.4 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc001gmi.4 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.17 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc001gmj.4 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.13 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc010pne.2 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc010pnf.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 1.17 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc010png.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 1.17 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (100) | 100.0 |
| 526 | ABL2 | 2212 | uc010pnh.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.6 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (100) | 100.0 |
- 7 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 526 | chr1 | 179068721 | 179068955 | wgEncodeRegDnaseClusteredV2 | 6 | score:185, srow:84865 |
| 526 | chr1 | 179068739 | 179068762 | tfbsConsSites | V$COMP1_01 | score:788, zScore:1.75, srow:373518 |
| 526 | chr1 | 179068744 | 179068757 | tfbsConsSites | V$CEBP_Q2 | score:889, zScore:1.86, srow:373519 |
| 526 | chr1 | 179068753 | 179068771 | tfbsConsSites | V$GR_Q6 | score:829, zScore:1.79, srow:373520 |
| 526 | chr1 | 179068844 | 179068853 | tfbsConsSites | V$TATA_C | score:894, zScore:1.83, srow:373521 |
| 526 | chr1 | 179068759 | 179068763 | phastConsElements100way | lod=19 | score:285, srow:643374 |
| 526 | chr1 | 179068787 | 179068796 | phastConsElements100way | lod=16 | score:268, srow:643375 |
| 526 | chr1 | 179068808 | 179068815 | phastConsElements100way | lod=17 | score:274, srow:643376 |
| 526 | chr1 | 179068819 | 179068833 | phastConsElements100way | lod=125 | score:471, srow:643377 |
| 526 | chr1 | 179068836 | 179068837 | phastConsElements100way | lod=13 | score:247, srow:643378 |
| 526 | chr1 | 179068867 | 179068888 | phastConsElements100way | lod=21 | score:295, srow:643379 |
- 11 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 526 | cellmeth_recounts | PrEC: 10, LNCaP: 1, DU145: 3 | 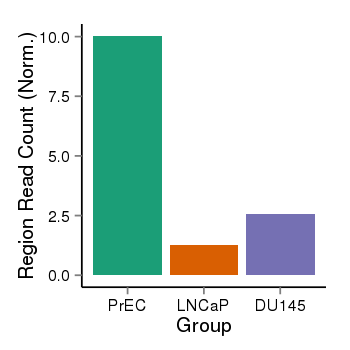 |
| 526 | cellexp | id=ENSG00000143322, name=ABL2, PrEC=5055, str=11904, LNCaP=1282, str=1641, DU145=11778, str=16871 | 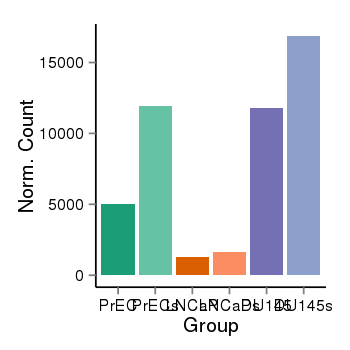 |
| 526 | tcgaexp | gene=ABL2, entrez=27, pos=chr1:179068462-179198819(-), B=1501, L=1642, M=1768, H=1925 | 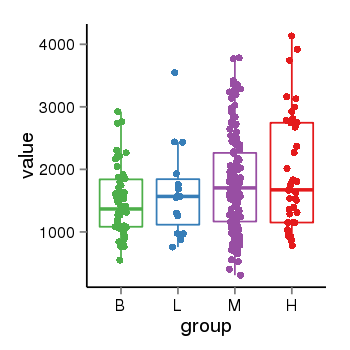 |
| 526 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |