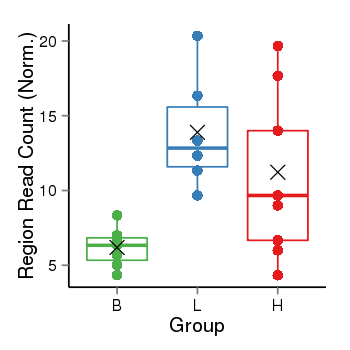
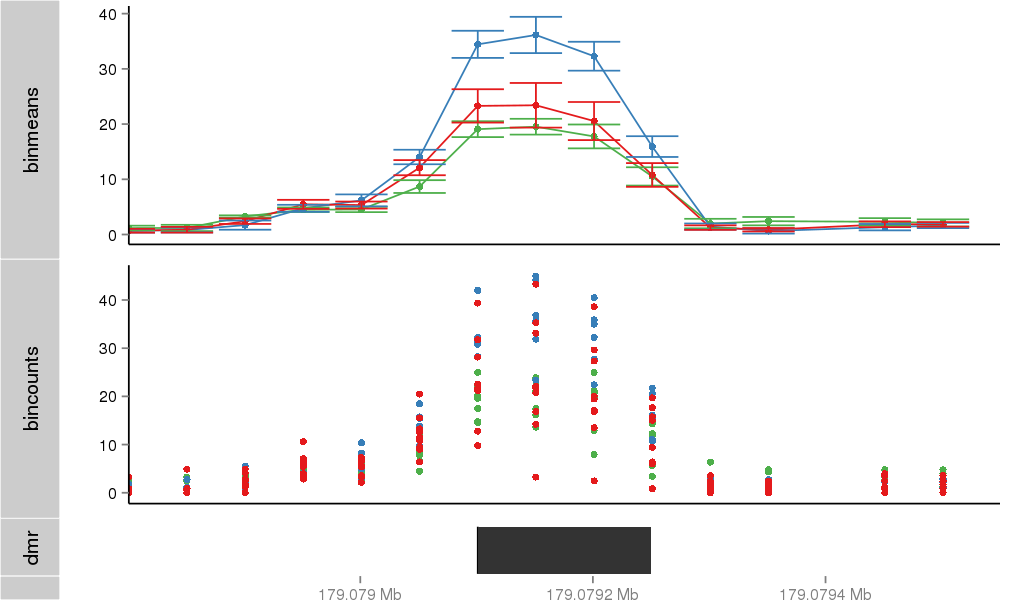
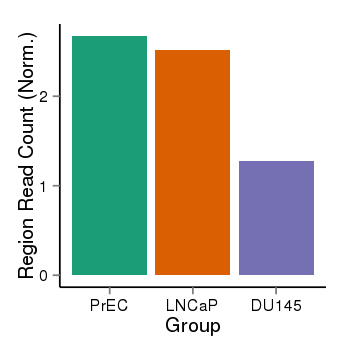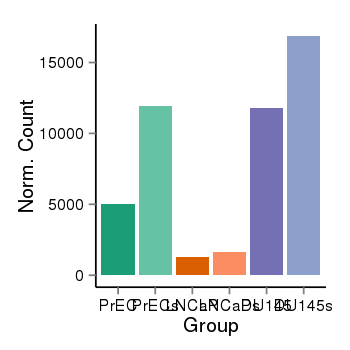| 527 | ABL2 | 2212 | uc001gmg.4 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.6 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (100), E12 (0), I12 (0), E13 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc001gmi.4 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (100), E12 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc001gmj.4 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.13 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (100), E12 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc010pne.2 | chr1 | 179068462 | 179112224 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (100), E11 (0), I11 (0), E12 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc010pnf.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.13 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (100), E12 (0), I12 (0), E13 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc010png.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (100), E11 (0), I11 (0), E12 (0) | 0.0 |
| 527 | ABL2 | 2212 | uc010pnh.2 | chr1 | 179068462 | 179198819 | - | 150 | 100.0 | 0.1 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (100), E11 (0) | 0.0 |