DMR 66 chr1:5950701-5951000 (300bp)
| DMR Stats | |
|---|---|
| Position | chr1:5950701-5951000 (View on UCSC) |
| Width | 300bp |
| Genomic Context | exon |
| Nearest Genes | NPHP4 (0bp away) |
| ANODEV p-value | 0.0105726550084 |
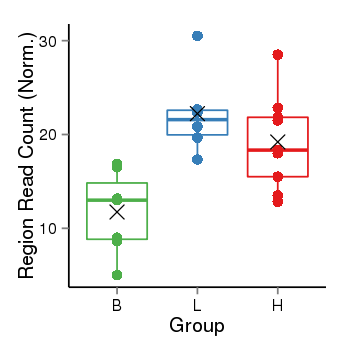
| Frequencies | Benign: 7.0 (100.0%), Low: 6.0 (100.0%), High: 9.0 (100.0 %) |
| Frequent? |
Region Count Boxplot
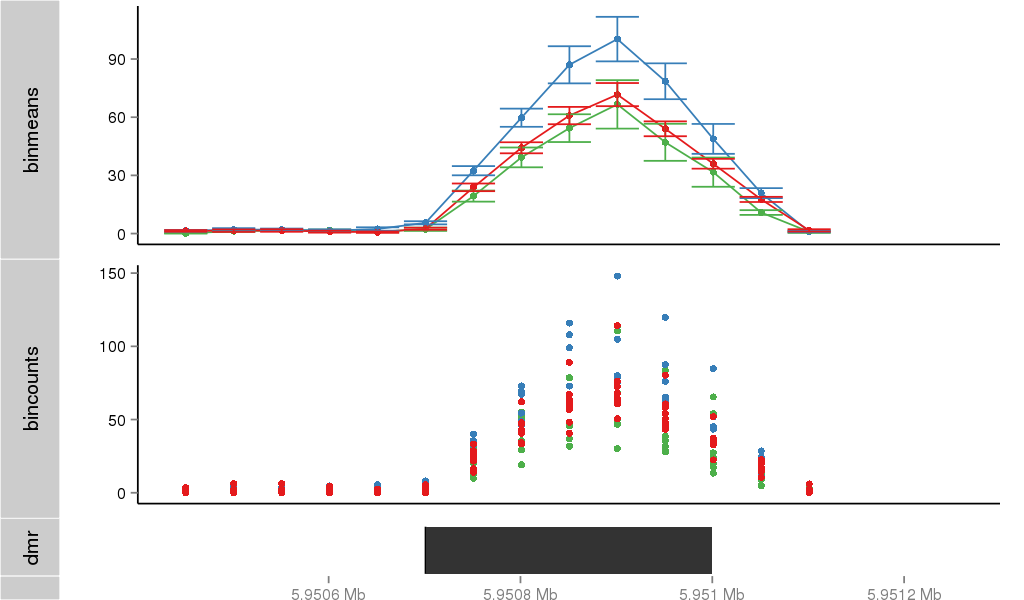
Region Overview
Gene Overlaps
| Dmrid | Gene Symbol | Gene Id | Isoform Id | Isoform Chr | Isoform Start | Isoform End | Isoform Strand | Overlap Bp | Query Overlap Per | Isoform Overlap Per | Noncoding | Promoter Per | Exonintron Per | End Per |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 66 | NPHP4 | 140 | uc001alq.2 | chr1 | 5922870 | 6052533 | - | 300 | 100.0 | 0.33 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (24.33), I17 (75.67), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0), I23 (0), E24 (0), I24 (0), E25 (0), I25 (0), E26 (0), I26 (0), E27 (0), I27 (0), E28 (0), I28 (0), E29 (0), I29 (0), E30 (0) | 0.0 |
| 66 | NPHP4 | 140 | uc001als.2 | chr1 | 5936014 | 6052533 | - | 300 | 100.0 | 0.18 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0), I16 (0), E17 (24.33), I17 (75.67), E18 (0), I18 (0), E19 (0), I19 (0), E20 (0), I20 (0), E21 (0), I21 (0), E22 (0), I22 (0), E23 (0) | 0.0 |
| 66 | NPHP4 | 140 | uc001alt.3 | chr1 | 5937153 | 6052533 | - | 300 | 100.0 | 0.18 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (24.33), I13 (75.67), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 66 | NPHP4 | 140 | uc009vlt.2 | chr1 | 5937153 | 6052533 | - | 300 | 100.0 | 0.24 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (24.33), I14 (75.67), E15 (0), I15 (0), E16 (0), I16 (0), E17 (0) | 0.0 |
| 66 | NPHP4 | 140 | uc009vlu.2 | chr1 | 5946555 | 5965543 | - | 300 | 100.0 | 0.18 | 1 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (0), E3 (24.33), I3 (75.67), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0) | 0.0 |
- 5 geness
Feature Overlaps
| Dmrid | Feature Chr | Feature Start | Feature End | Set | Name | Fields |
|---|---|---|---|---|---|---|
| 66 | chr1 | 5950866 | 5951030 | wgEncodeRegDnaseClusteredV2 | 2 | score:299, srow:4428 |
| 66 | chr1 | 5950975 | 5950988 | tfbsConsSites | V$ATF_01 | score:852, zScore:2.17, srow:8847 |
| 66 | chr1 | 5950973 | 5950973 | cosmic | COSM911164 | srow:2862 |
| 66 | chr1 | 5950925 | 5950932 | phastConsElements100way | lod=102 | score:451, srow:20221 |
| 66 | chr1 | 5950938 | 5950939 | phastConsElements100way | lod=17 | score:274, srow:20222 |
| 66 | chr1 | 5950941 | 5950942 | phastConsElements100way | lod=18 | score:280, srow:20223 |
| 66 | chr1 | 5950944 | 5950944 | phastConsElements100way | lod=15 | score:262, srow:20224 |
| 66 | chr1 | 5950947 | 5950948 | phastConsElements100way | lod=36 | score:348, srow:20225 |
| 66 | chr1 | 5950950 | 5950951 | phastConsElements100way | lod=28 | score:323, srow:20226 |
| 66 | chr1 | 5950953 | 5950953 | phastConsElements100way | lod=16 | score:268, srow:20227 |
| 66 | chr1 | 5950956 | 5950957 | phastConsElements100way | lod=34 | score:343, srow:20228 |
| 66 | chr1 | 5950959 | 5950962 | phastConsElements100way | lod=62 | score:402, srow:20229 |
| 66 | chr1 | 5950965 | 5950969 | phastConsElements100way | lod=52 | score:385, srow:20230 |
| 66 | chr1 | 5950971 | 5950972 | phastConsElements100way | lod=19 | score:285, srow:20231 |
| 66 | chr1 | 5950974 | 5950980 | phastConsElements100way | lod=96 | score:445, srow:20232 |
| 66 | chr1 | 5950983 | 5950984 | phastConsElements100way | lod=31 | score:333, srow:20233 |
| 66 | chr1 | 5950986 | 5950999 | phastConsElements100way | lod=111 | score:460, srow:20234 |
| 66 | chr1 | 5950825 | 5951029 | cpgIsland | CpG: 16 | length:205, cpgNum:16, gcNum:131, perCpg:15.6, perGc:63.9, obsExp:0.77, srow:306 |
| 66 | chr1 | 5948825 | 5950824 | cpgShore | — | values(x.gr)[overs$srow, ]:509 |
- 19 featuress
Omics Overlaps
| Dmrid | Omic Set | Data | Plot |
|---|---|---|---|
| 66 | cellmeth_recounts | PrEC: 61, LNCaP: 6, DU145: 6 | 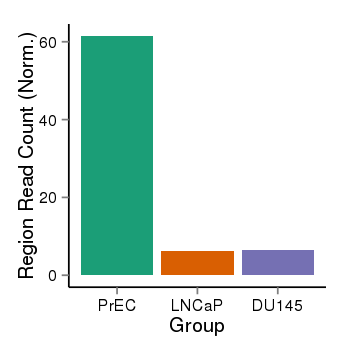 |
| 66 | cellmeth_diffreps_PrECvsLNCaP | A=95, B=5, Length=520, Event=Down, log2FC=-4.25, padj=0 | 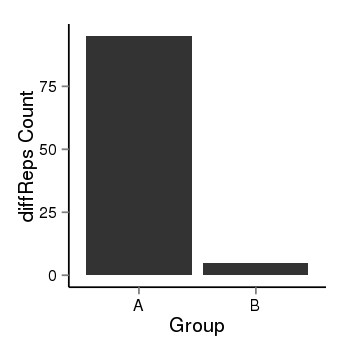 |
| 66 | cellmeth_diffreps_PrECvsDU145 | A=95, B=4, Length=500, Event=Down, log2FC=-4.57, padj=0 | 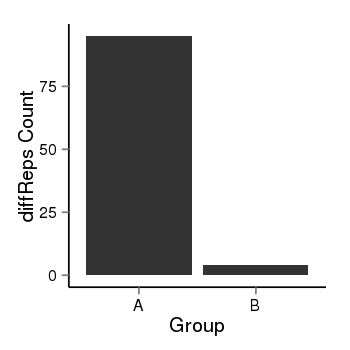 |
| 66 | cellexp | id=ENSG00000131697, name=NPHP4, PrEC=1480, str=1194, LNCaP=1452, str=836, DU145=1085, str=983 | 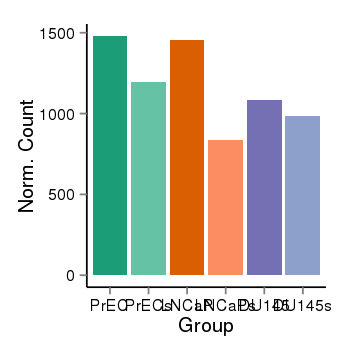 |
| 66 | tcgameth | site:cg16164843, B=0.88, L=0.89, H=0.89 | 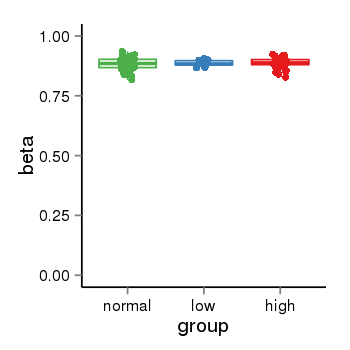 |
| 66 | tcgameth | site:cg22399023, B=0.86, L=0.92, H=0.91 | 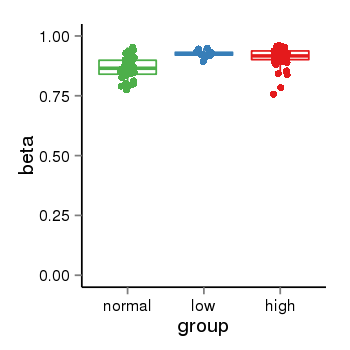 |
| 66 | tcgaexp | gene=SRSF10, entrez=10772, pos=chr1:36273-24306953(-), B=3487, L=3388, M=3432, H=4019 | 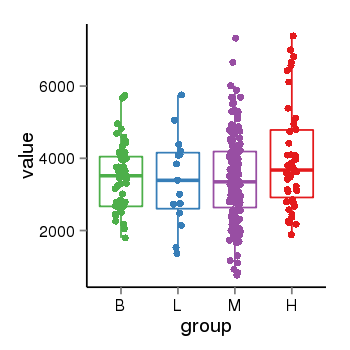 |
| 66 | tcgaexp | gene=NPHP4, entrez=261734, pos=chr1:5922870-6052533(-), B=469, L=561, M=543, H=654 | 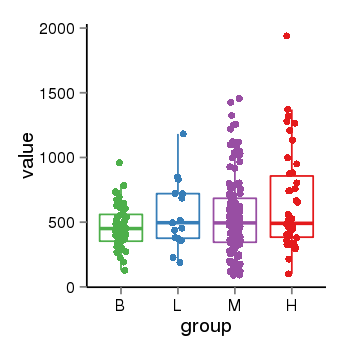 |
| 66 | tcgaexp | gene=LOC100133331, entrez=100133331, pos=chr1:322037-180753553(-), B=268, L=328, M=271, H=305 |  |