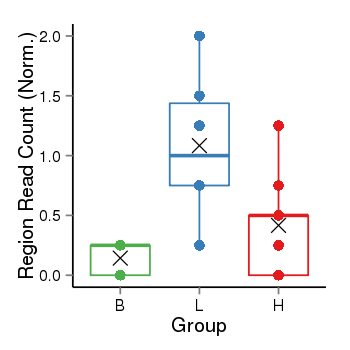
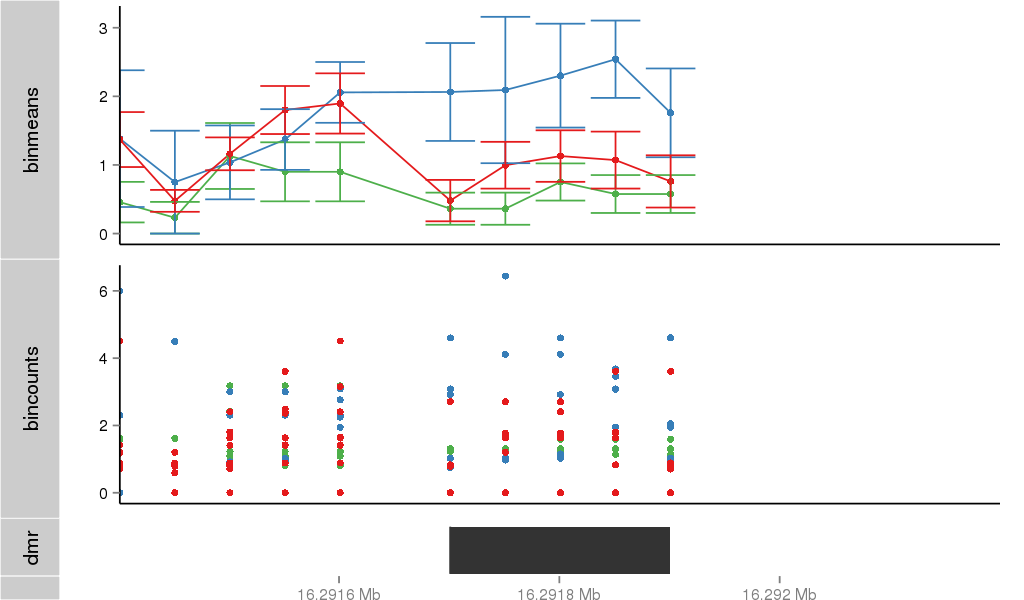
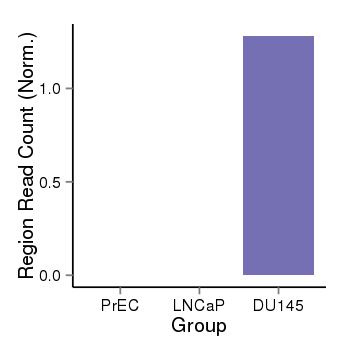
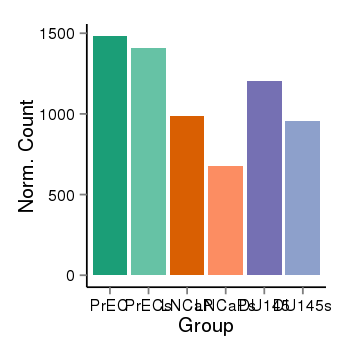| 137 | ZBTB17 | 288 | uc001axl.4 | chr1 | 16268364 | 16302627 | - | 200 | 100.0 | 0.05 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc009vom.1 | chr1 | 16271432 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc009von.1 | chr1 | 16274238 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (100), E2 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obq.2 | chr1 | 16268364 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obr.2 | chr1 | 16268364 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obs.2 | chr1 | 16268364 | 16302627 | - | 200 | 100.0 | 0.05 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0), I8 (0), E9 (0), I9 (0), E10 (0), I10 (0), E11 (0), I11 (0), E12 (0), I12 (0), E13 (0), I13 (0), E14 (0), I14 (0), E15 (0), I15 (0), E16 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obt.1 | chr1 | 16270322 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0), I7 (0), E8 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obu.2 | chr1 | 16270365 | 16302627 | - | 200 | 100.0 | 0.04 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0), I5 (0), E6 (0), I6 (0), E7 (0) | 0.0 |
| 137 | ZBTB17 | 288 | uc010obv.1 | chr1 | 16272257 | 16302627 | - | 200 | 100.0 | 0.12 | 0 | 0.0 | E1 (0), I1 (0), E2 (0), I2 (100), E3 (0), I3 (0), E4 (0), I4 (0), E5 (0) | 0.0 |